August Krogh Building Copenhagen University. Universitetsparken Copenhagen OE
|
|
|
- Agnete Brandt
- 8 år siden
- Visninger:
Transkript
1 Per Amstrup Pedersen Institute of Molecular Biology August Krogh Building Copenhagen University Universitetsparken Copenhagen OE En genteknologisk værktøjskasse. Version 2007
2 Indholdsfortegnelse Southern blot 2 3 Northern blot 3-4 Western blot 4 5 Immunoprecipitation 4, 6 RNase protection 6 7 Primer ekstension 8 Bandshift (EMSA) 9 Colony/plaque lift S1 nuklease mapning 10, 12,13 Mærkning af prober 10, 14, 15 Nuclear run on 15, 16 5 RACE RACE 19 Inverse PCR 20 En hybrid system To hybrid system Polysomanalyse 25, 26, 27 Filterbindingsassay Site directed mutagenese Fusion af to DNA fragmenter 34 Proteomics Transkriptom analyse Differential display Expressionskloning Pulse-chase forsøg Real time PCR
3 Følgende katalog er tænkt som en præsentation af en række vigtige molekylærbiologiske teknikker, som ikke er beskrevet direkte i lærebogen. Anvendelse af teknikkerne til løsning af konkrete molekylærbiologiske problemstillinger vil blive illustreret i forbindelse med opgave diskussionerne, men for at lette forståelsen beskrives principperne kort i dette kompendium. Nukleinsyre hybridisering. En af de vigtiske teknikker til påvisning af et givent DNA eller RNA molekyle i et biologisk materiale bygger på komplementære nukleinsyrers evne til at binde til hinanden via hydrogenbindinger. Denne proces benævnes hybridisering. Hybridiseringen kan enten foregå i opløsning eller på en membran. Langt de fleste hybridiseringen foretages på en membran. For at visualisere hybridiseringsproduktet anvendes en mærket probe. Mærkningen kan enten foretages ved at inkorporere et eller flere radioaktive nukleotider i proben eller ved at inkorporere biotinylerede nukleotider i proben. Lange prober fremstilles enten ved primer ekstension, nick translation, eller random labelling medens kortere, biotinylerede prober kan fremstilles direkte på DNA syntesemaskinen. Begge typer mærkning bevirker at hybridiseringsproduktet kan visualiseres ved at lægge en røntgen film over hybridiseringblandingen eller ved PhosphorImage analyse. PhosphorImage analyse foregår ved, at den det radioaktivt mærkede materiale lægges ned i en phosphorimage kasette med en PhosphorImage plade, som er i stand til at absorbere β-stråling. Molekyler i pladen exiteres af β-strålingen og den absorberede energi frigives først, når pladen scannes af en laser i PhosphorImage maskinen. På dette tidspunkt opsamles og kvantificeres den frigivne energi af en monitor og en computer regenererer et billede. Fordelen ved PhosphorImage analyse er, at kvantificeringen er mere nøjaktig end den, der kan opnås ved almindelig autoradiografi på røntgenfilm. Problemet med filmen er, at sammenhængen mellem sværtning af filmen og radioaktiviteten i prøven kun er lineær over et forholdsvis lille område. Southern blotting. Den oprindelige metode til blotting blev udviklet af Southern i Metoden bygger på at restriktionsskåret DNA separeres i en agarosegel. Herefter blottes DNAet over på en nylon eller nitrocellulose membran. Dette blev i den oprindelige protokol gjort ved kapilært flow men gøres i dag oftest elektroforetisk eller med sug eller tryk. Hermed reduceres overførselstiden fra overnat til 1 time. DNA bindes til membranen enten ved bagning eller ved bestråling med UV lys. For at overføre DNAet effektivt fra gel til membran, og for at sikre at alt DNA overføres lige effektivt, er det nødvendigt at fragmentere DNAet. Dette gøres ved at inkubere agarosegelen i 0,25M HCl. Dette depurinerer DNAet og gør fosfordiesterbindingen tilgængelig over for efterfølgende spaltning med NaOH. Efter overførsel af DNAet til membranen inkuberes denne med den mærkede denaturerede probe, som så vil binde til de DNA molekyler på filteret, der har tilstrækkelig sekvenshomologi til proben. Den nødvendige grad af sekvenshomologi afhænger af, hvor stringente hybridiserings-betingelserne er. Her er temperaturen og saltkoncentrationen vigtige parametre, idet der kræves højere sekvenshomologi for at proben kan binde, jo højere hybridiseringstemperaturen er. Samtidig kræver hybridisering i lav salt højere sekvenshomologi end hybridisering i høj salt. Positionen i gelen af det fragment, der 2
4 har sekvenshomologi til proben, visualiseres ved autoradiografi på en røntgenfilm eller med en PhosphoImager. De enkelte trin i Southern blotting er vist i Figur 1. Separer skåret DNA ved agarose gelelektroforese Figur 1: Princippet i Southern blotting teknikken. Restriktionsskåret DNA separeres ved agarose-gel-elektroforese. Efter depurinering med HCl og spaltning med NaOH overføres DNAet til en membran. Efter endt overførsel inkuberes membranen i et lille volume hybridiseringsvæske sammen med en mærket probe, hvis sekvens er komplementær til det stykke DNA man gerne vil visualisere. For at kunne bestemme størrelsen af DNA fragmenterne fotograferes den ethidiumbromid farved gel sammen med en lineal før overførslen. 3
5 Northern blotting. Proceduren i northern blotting er i princippet den samme som for Southern blotting blot kan vi påvise tilstedværelsen af et bestemt RNA molekyle i et RNA ekstrakt. RNA separeres i modsætning til DNA i formamid holdige agarosegeler for at holde RNAet denatureret under gelkørslen. RNAet overføres til en nylon membran eller en nitrocellulose membran, bages eller UV bestråles og tilsættes den mærkede probe. Efter hybridisering visualiseres positionen af det til proben komplementære RNA molekyle ved autoradiografi eller med en PhosphoImager. Som probe kan anvendes både DNA og RNA prober. RNA-RNA hybrider har højere smeltetemperatur end DNA-RNA prober. Western blotting. Efter udvikling af Southern og northern blotting teknikkerne blev der udviklet en analog teknik, western blotting, til påvisning af et bestem protein i et protein ekstrakt. Proteinerne i ekstraktet separeres i en SDS holdig polyakrylamid gel og overføres til en nitrocellulose membran eller en nylon membran. Som probe anvendes et antistof rettet mod det protein, vi vil påvise tilstedeværelsen af. Dette primære antistof vil binde til det på membranen immobiliserede protein. Ved anvendelse af et enzym-mærket eller radioaktiv mærket sekundært antistof kan det primære anstistofantigen kompleks visualiseres ved tilsætning af enzymsubstratet,ved autoradiografi. eller ved PhosphoImage analyse. Princippet i western blot proceduren er vist i Figur 2. Immunoprecipitation. Som en alternativ metode til identifikation af et bestemt protein i et protein ekstrakt kan anvendes immunoprecipitation. Dette gøres som oftest efter, at proteinerne i en cellekultur er blevet radioaktiv mærket efter tilsætning af radioaktive aminosyrer til vækstmediet. Dette er som oftest S, C eller 3 H mærkede aminosyrer. Cellerne lyseres og inkuberes med et IgG antistof rettet mod det protein, der ønskes identificeret. Efter antigen-antistof komplekset er dannet tilsættes enten døde Staphyllococcus aureus celler eller sepharose kugler coated med protein A eller protein G. Fidusen er, at protein A og protein G binder til Fc delen af IgG antistoffer. S. aureus celler indeholder netop protein A og G proteiner på overfladen. Resultatet er, at vi får dannet et antistof-antigen-protein A/G-sepharose kugle kompleks, som kan isoleres fra resten af cellens proteiner ved centrifugering. Når dette kompleks loades på en denaturerende poly-akryl-amid-gel (SDS-PAGE) går komplekset i stykker og de enkelte proteiner separeres i gelen. Da det kun er det immunpræcipiterede protein, som er radioaktiv mærket, er det kun dette protein, der visualiseres ved autoradiografi eller PhosphoImage analyse. Princippet i Immunoprecipitations-proceduren er vist i Figur 3. 4
6 MW MW MW MW MW. Figur 2: Western blotting. De enkelte trin i western blotting proceduren til påvisning af et bestemt protein i et proteinekstrakt. Teknikken kan også bruges til at kvantificere mængden af ef et givent antigen, hvis man kan fremstille en standardkurve ud fra forskellige mængder påsat oprenset antigen. MW angiver en molekylvægtsmarkør, så størrelse af det identificerede antigen kan bestemmes. 5
7 Radioaktiv mærket protein ekstrakt inkuberes i opløsning med antistof Tilsæt protein A/G coatede sepharose kugler eller inaktiverede Staphyllococcus aureus celler Sedimenter i centrifugen og kasser supernatanten Separer immunpræcipitatet ved SDS-PAGE og visualiser proteinbåndede ved autoradiografi eller på PhosphoImager Figur 3: Skematisk oversigt over immunoprecipitationsproceduren. Forskellige proteiner er angivet med forskellige farver. Sepharosekugler/S. aureus celler er angivet med grønt. RNase protektion assay. Dette er et alternativ til northern blotting og er mere sensitivt. Princippet er, at en fremstillet radioaktiv probe indkuberes med den RNA preparation, der ønskes undersøgt. Er der RNA molekyler med sekvenser, der er komplementære til proben, vil der dannes en dobbeltstrenget, mærket RNA-probe hybrid. Dette kompleks vil i modsætning til det ikke-hybridiserede RNA være resistent overfor RNase A behandling. Tilsættes der derfor et overskud af probe, vil alle RNA molekyler med sekvenser komplementære til proben ikke blive fordøjet helt. Kun den del af RNA 6
8 molekylet, der ikke hybridiserer med proben, vil blive nedbrudt. Separation af det RNase fordøjede RNA i en formamid-holdig polyacrylamidgel efterfulgt af autoradiografi eller PhosphoImage analyse viser et bånd, der svarer til længden af proben. Princippet i RNase protection teknikken er vist i Figur 4. Isoler mrna fra det interessante væv. mrna mrna mrna mrna mrna mrna Tilsæt overskud af radioaktiv mærket, fremstillet RNA. AAAAA AAAAA AAAAA in vitro AAAAA AAAAA AAAAA Inkuber med RNase A som fjerner enkeltstrenget RNA A B C Lane A: RNA + probe brugt til forsøget Lane B: kun RNA brugt til forsøget Lane C: kun probe brugt til forsøget Figur 4: Princippet i RNase protection analyse. Efter hybridisering med en mærket RNA probe (angivet i rødt og gult), tilsættes RNase A som nedbryder ikke hybridiseret RNA. Efter elektroforese i en denaturerende poly-acryl-amid-gel og autoradiografi/phosphorimage analyse vil mængden af radioaktivitet i det fremkomne bånd reflektere mængden af mrna, der var i den isolerede prøve. Som negative kontroller laves de under B og C benævnte forsøg. 7
9 Primer ekstention. Primer ekstention en fællesbetegnelsen for en teknik, der involver hybridisering af en primer til et stykke komplemetært nukleinsyre efterfulgt af forlængelse af primeren med en polymerase. Teknikken bruges især til mapning af transkriptions startsitet for promotorer, d.v.s. identifikation af hvilket nukleotid, der er det første i 5 enden af et mrna molekyle. Teknikken kræver, at genet der koder for mrna molekylet er kendt og at exon-intron strukturen er kendt. Primerekstension teknikken er vist i Figur 5. Isoler mrna (evt. totalt RNA) fra det ønskede væv mrna 5 AAAAA 3 mrna 5 RT-primer 3 5 Anneal den kinerede RT-primer til mrnaet mindre end 100 baser fra den formodede 5' ende. AAAAA 3 Tilsæt reverse transkriptase og nukleotider mrna 3 5 RT-primer 5 Sekventer en genomklon med samme primer og separer primerekstension produktet og sekvensprodukterne på den samme denaturerende polyacrylamidgel AAAAA 3 P G A T C Identificer det bånd i sekvensreaktionen der har samme længde som primerekstension produktet og bestem længden på dette. Dette kan gøres idet vi. har kendskab til nukleotidsekvensen af det sekventerede DNA og den anvendte primer. Sekvensreaktionerne fungerer i dette tilfælde fungerer som lineal på for hvor lang primer-ekstension-produktet er. Er primer-ekstension-produktet f.eks. 55 nukleotider langt kan vi gå ind i sekvensen af det genomiske DNA hvorfra mrnaet blev transkriberet og tælle 55 nukleotider fra 5' enden af den til forsøget anvendte primer.her findes transkriptions-startsitet. Figur 5: Illustration af primerekstension teknikken. Teknikken er baseret på, at en sekvensspecifik, radioaktiv mærket primer anneales til et område i 5 enden af mrna og forlænges ved hjælp af en reverse transkriptase indtil 5 enden af mrna molekylet. Lægden af primerekstension produktet kan dernæst bestemmes efter elektroforese i en urinstofholdig denaturerende polyacrylamidgel med et sæt sekvensreaktioner som lineal. 8
10 Band shift assay, EMSA (electrophoretic mobility shift assay) Denne teknik bruges oftest til at undersøge, om der i cellekernen findes et eller flere proteiner, der specifikt binder til promoter regionen af et gen. Der skal anvendes radioaktivt mærkede oligonukleotider, der dækker forskellige dele af promotoren og et protein ekstrakt fra kerner (kerne ekstrakt) fra de celler, man arbejder med. Alternativt/suplerende kan er anvendes rekombinant fremstillet og oprenset protein. Blandes kerneekstraktet/oprenset protein med radioaktive oligonukleotider efterfulgt af separation i en ikke-denaturerende polyacrylamidgel og autoradiografi eller PhophoImage analyse, vil de nøgne oligonukleotider have en højere mobilitet i gelen end oligonukleotid-protein komplekset. Positionen af oligonukleotidet i gelen bliver således shiftet når der er protein i kerneekstraktet, der har affinitet for oligonukleotidet. Proteinerne, der binder, vil som oftes være transkritionsfaktorer. Har man antistoffer mod forskellige transkriptionsfaktorer, kan man undersøge om tilsætning af disse vil forårsage et supershift af positionen af oligonukleotid-protein komplekset. På denne måde kan et EMSA forsøg identificere de DNA bindende proteiner. Assayet kan også udføres med RNA. Princippet i et EMSA assay er vist i Figur 6. A B C Supershift Shift Nøgent oligonukleotid Figur 6: Illustration af EMSA teknikken. Radioaktivt mærkede dobbeltstrengede DNA molekyler blandes med kerekstrakt og blandingene separeres i en ikke-denaturerende poly-acryl-amidgel. Hvis der binde protein til DNA molekylerne får disse en lavere mobilitet i gelen. Tilsættes yderlige antistof mod det DNA bindende protein før separation i gelen fås et supershift, idet mobiliteten nedsættes yderliger. A, kun tilsat oligonukleotider; B, tilsat oligonukleotider og kerneekstrakt; C, tilsat, oligonukleotider, kerneekstrakt og antistof. 9
11 Identifikation af enkelte kolonier der indeholder et bestemt stykke DNA eller protein. Hybridiseringsteknikkerne kan også bruges i forbindelse med screening af genbiblioteker. Hele kolonier kan føres over på et membranfilter (man taler om colony lift) og efter lysering af cellerne kan DNAet eller proteinet bindes til membranen. Har man en mærket probe, kan man som i Southern Blotting teknikken identificere kolonier, der indeholder nuklein syre sekvenser, der er komplementære til proben. Helt analogt kan man, hvis man har et antistof, identificere de kolonier, der udtrykker et bestemt protein. Selve visualiseringen af de rigtige kolonier foregår som under western blotting. Ofte bruges teknikken til at identificere lambda fager indeholdende sekvenser komplementære til en probe. Dette gør sig gældende ved screening af genom og cdna biblioteker. Her taler man om plaque lift idet de plaques som dannes efter lambda infektion løftes over på et membranfilter. Teknikken er illustreret i Figur 7. Nuklease S1 mapning af transkriptions start sites. Dette er et alternativ til primer ekstenstion teknikken. Teknikken kræver at nukleotid sekvensen af genet er kendt. Princippet minder om princippet i RNase protection. Et radioaktiv mærket nukleotid hybridiseres til RNA efterfulgt af fordøjelse med nuklease S1 som fjerner enkeltstrenget nukleinsyre. Da 5 enden af den mærkede oligo kendes, kan man ved at bestemme længden af det ikke-fordøjede oligo nukleotid bestemme, hvor mange nukleotider der er fra oligoens 5 ende til det første nukleotid der transkiberes af RNA-polymerasen. Dette er netop 5 enden af mrnaet. Ved i genomsekvensen at tælle det bestemte antal nukleotider op fra oligoens 5 ende (introns springes over), findes det første nukleotid der transkriberes. Teknikken er illustreret i Figur 8. Teknikken kan helt analogt anvendes til at bestemme 3 enden af mrna. Dette er vist i Figur 9. Mærkning af prober. Nick translation bygger på at DNase I kan lave et nick (bryde fosfordiesterbindingen) i den ene streng af et dobbeltstrenget DNA molekyle. DNase I mængden titreres således, at hvert DNA molekyle kun får lavet et nick. Tilstedeværelse af DNA polymerase vil bevirke at dennes 5-3 exonuklease aktivitet vil fjerne nukleotider startende fra det område, hvor DNasen har lavet et nick. Samtidig vil 3 enden fra det sted, hvor nicket er sket, kunne bruges af DNA polymerasen som primer til nysyntese af DNA med den komplementære streng som template. Sker syntesen i tilstedeværelse af radioaktive nukleotider vil replikationsproduktet blive radioaktiv. Random labeling bygger på primer ekstension teknikken idet en blanding af alle mulige hexanukleotider hybridiseres med det DNA fragment, der skal mærkes. Tilsætning af en DNA polymerase og mærkede nukleotider forlænger primeren og inkorporerer de mærkede nukleotider. I stedet for at anvende en blanding af alle hexanukleotider, kan der i stedet anvendes en sekvens specifik primer. Teknikkerne er illustreret i figur 10 og figur
12 Petrisklål med bakterie kolonier eller λ-plaques (masterplade) Overfør bakterier til membran (kaldes colony lift henholdsvis plaque lift på engelsk) Lyser cellerne på filteret hvis plaques er dette trin overflødigt Inkuber med mærket probe ( ) Fremkald som ved Southern blot Inkuber med antistof ( ) Fremkald som ved western blot Figur 7. Princippet i colony lift henholdsvis plaque lift. 11
13 mrna 5' mrna 5' AAAAA Anneal en radioaktiv mærket sekvensspecifik primer til mrna AAAAA Tilsæt nuklease S1 mrna 5' Separer produktet i en denaturerende poly-acryl-amid-gel sammen med en sekvensreaktion S1 G A T C Figur 8: Bestemmelse af transkriptionsstartsite med S1 nukelase behandling. En sekvensspecifik, radioaktiv mærket primer der når udover mrnaets 5 ende anneales. Herefter fjerner nuklease S1 alt det enkeltstrengede DNA og RNA. Det, der bliver tilbage, er således den del af den mærkede probe, der når hen til 5 enden af mrnaet. Længden af den tilbageværende del af proben kan bestemmes efter elektroforese i en denaturerende poly-acryl-amid-gel sammen med en sekvensreaktion, der fungerer som lineal. Måles fragmentlængden f.eks. til 58 nukleotider kan vi gå ind i DNA sekvensen for det pågældende gen og tælle 58 nukleotider op fra 5 enden af den radioaktiv mærkede primer. Dette nukleotid er det første der transkriberes. 12
14 mrna 5' AAAAA Anneal en radioaktiv mærket sekvensspecifik primer til mrna mrna 5' AAAAA Tilsæt nuklease S1 mrna 5' Separer produktet i en denaturerende poly-acryl-amid-gel sammen med en sekvensreaktion S1 G A T C Figur 9: Bestemmelse af 3 enden af mrna ved S1-nuklease mærkning. En sekvensspecifik, radioaktiv mærket primer der når udover mrnaets 3 ende anneales. Herefter fjerner nuklease S1 alt det enkeltstrengede DNA og RNA. Det, der bliver tilbage, er således den del af den mærkede probe, der når hen til 3 enden af mrnaet. Længden af den tilbageværende del af proben kan bestemmes efter elektroforese i en denaturerende poly-acryl-amid-gel sammen med en sekvensreaktion, der fungerer som lineal. Måles fragmentlængden f.eks. til 58 nukleotider kan vi gå ind i DNA sekvensen for det pågældende gen og tælle 58 nukleotider ned fra 5 enden af den radioaktiv mærkede primer. Dette nukleotid svarer til det sidste i det færdige mrna. 13
15 Før brug testes proben for om den er mærket tilstrækkeligt. Dette kan gøres ved at separere proben fra u-indkorporerede nukleotider på en søjle. Umiddelbart før brug koges proben for at den skal denaturere. Figur 10: Mærkning af prober med nick-translation. 14
16 Her benyttes principppet i primerekstension, idet der som primerer anvendes en blanding af alle mulige hexamerer ( ). Denaturer DNAet og tilsæt tilfældige hexamerer Tilsæt DNA polymerase og mærkede nukleotider (kan være biotinylerede eller radioaktive)( ) Resultatet er stykker af mærket DNA. Figur 11: Mærkning af prober med random hexamerer. Nuclear run on. Denne teknik anvendes til sammenligning af transkriptions-initieringsfrekvensen af et givet gen under forskellige betingelser. Der isoleres kerner fra de celler, der arbejdes med og der tilsættes en inhibitor af transkriptionsinitieringen samt radioaktive nukleotider. Disse vil blive indkorporeret i det RNA, der var under syntese da kernerne blev isoleret. Under antagelse af at RNA nedbrydningen i kernen er negligeabel, vil den mængde RNA, der dannes i et givent tidsrum, afhænge af hvor mange RNA polymeraser, der på kerne isolerings tidspunktet var i gang med at transkribere. Det RNA transkript man gerne vil påvise kan identificeres ved northern blotting eller dot blotting med en mærket probe. Teknikker er illustreret i figur 12 15
17 Isoler kerner fra de celler der skal undersøges og tilsæt inhibitorer af transkriptions-initieringen og nukleotider. Umiddelbart efter inhibering af initieringen får den sidste RNA polymerase således lov at initiere transkriptionen, medens de, der er i gang, får lov at "køre" færdig. Den sidste RNA polymerase der får lov at initiere Transkriptionen forløber indtil den sidste RNA polymerase terminerer transkriptionen Tilstedeværelse og kvantificering af de enkelte transkripter kan ske med med northern blotting, RNase protection eller slot blotting. Figur 12: Illustration af nuclear run on teknikken. 16
18 5 RACE (Rapid Aplification of cdna Ends) 5 RACE er en metode til at amplificere 5 enden af et stykke cdna. Ofte resulterer screening af et cdna bibliotek med en probe i isolering af en klon der mangler større eller mindre dele af 5 enden. I stedet for at konstruere et nyt bibliotek er det nemmere at bruge RT-PCR. RT reaktionen udføres med en primer der annealer til et område så tæt på mrnaets 5 ende som muligt (svarende til 5 enden af cdna sekvensen). Ved hjælp af terminal transferase påsættes der enten en polydg eller polydc hale på RT produktets 3 ende. Ved at udføre PCR med en polydc eller polydg primer samt den primer der blev brugt til RT rekationen får vi amplificeret den manglende 5 ende. Denne kan nu klones i et plasmid og nukleotidsekvensen kan bestemmes. Teknikken er illustreret i figur
19 mrna cdna mrna 5' AAAA 3' A Anneal den sekvensspecifike primer A og foretag reverse transkription 5' AAAA 3' Terminal transferase + dctp eller dgtp CCCCCC/ GGGGGG cdna mrna A 5' AAAA 3' PCR med primer A + polydc primer eller polydg primer Figur 13: Princippet i 5 RACE teknikken. 18
20 3 RACE 3 RACE er helt analogt til 5 RACE blot ønsker vi at få kendskab til 3 enden af et mrna molekyle. Til dette formål udfører vi en RT reaktion med en polydt primer, hvorpå der i 5 enden er påhæftet en hale med en kendt nukleotidsekvens. PCR med en halespecifik primer og en primer i det kendte stykke cdna efterfulgt af kloning og sekventering vil give nukleotidsekvensen af den ukendte 3 del af mrna molekylet. 3 RACE teknikken er illustreret i Figur 14. mrna cdna mrna cdna mrna 5' AAAA 3' Reverse transkription med polydt primer med halesekvensen A polydt A 5' AAAA 3' Anneal den sekvensspecifikke primer B til cdnaet B 5' AAAA 3' A PCR med primer A + primer B Figur 14: Illustration af 3 RACE teknikken. 19
21 Inverse PCR Ofte står man i den situation, at man har klonet og sekventeret et stykke kromosomal DNA, men mangler noget sekvens opstrøms og eller nedenstrøms for det sekventerede stykke DNA. For at tilvejebringe dette DNA kan man benytte sig af inversepcr. Konceptet bag denne teknik er vist i Figur ss. Figur 15: Skematisk oversigt over inverse PCR teknikken. Vi forudsætter at vi kender sekvensen af det røde område. Vi kan derfor fremstille 2 PCR primere som er specifikke for området og som har de viste orienteringer. Restriktionsanalyse af vores røde region viser at der ingen BamHI og PstI sites er. Derfor kan vi skære en portion kromosomal DNA med det ene enzym og et med det andet enzym uden at vi skærer i det røde område. Ligerer vi nu de mange fragmenter der genereres ved PstI eller BamHI skæring ved en meget lav koncentration af DNA vil fragmenterne langt overvejende ligere med sig selv. Vi får derfor genereret en række cirkulære molekyler af forskellig længde. Fidusen er, at kun et cirkulært DNA molekyle indeholdende det røde område kan PCR amplificeres med vores primere. Resultatet at PRC reaktionen er et linerært stykke DNA, der indeholder de ønskede opstrøms og nedenstrøm sekvenser. 20
22 Én-hybrid teknikken En-hybrid metoden anvendes ofte hvis man vil klone transkriptionsfaktorer fra højere eukaryoter. Transkriptionsfaktorer er karakteriseret ved at de kan binde en bestemt DNA sekvens (promotor element) og aktivere transkription. Problemet er imidlertid, at et transkriptionsaktiverende domæne fra højere eukaryoter ikke fungerer i gær. Så selv om trankriptionsfaktoren binder til den rigtige sekvens i en gærcelle, vil det transkriptionsaktiverende domæne ikke kunne interagere med det generelle transkriptionsapparat og aktivere transkriptionsinitiering. For at finde cdna kodende for den trankriptionsfaktor, der binder et bestemt identificeret DNA element, fremstiller vi et reporterplasmid med en minimalpromotor (TATA box) og det promotorelement, der bindes af vores ukendte transkriptionsfaktor. Samtidig fremstiller vi et cdna bibliotek Det transkriptionsaktiverende domæne af GAL4 (GAL4 TR ) fusioneres in frame til vores cdna. Det rigtige fusionsprotein vil binde til DNA elementet i vores reporterkonstruktion og dermed via GAL4 TR aktivere transkriptionen. Er ekspression af vores reportergen essentiel for gærens overlevelse vil kun de gærceller, der indeholder cdna, der koder for et protein med affinitet for vores DNA element overleve. Teknikken er illustreret i Figur 16 21
23 P TR GAL4 -cdna fusion GAL4 TR cdna GAL4 TR cdna DNA element Reportergen Figur 16: llustration af principperne bag en-hybrid teknikken. Der fremstilles et cdna bibliotek i en gærvektor indeholdende en konstitutiv promotor. cdna biblioteket transformeres i en gærstamme, der indeholder et reporterplasmid bestående af et DNA element fusioneret til en minimalpromotor og et reportergen, som f.eks. kunne være et LYS2 gen. Efter transformation i en lys2 stamme og selektion for lysin autotrofi vil kun transformanter, der er i stand til at initiere transkription af reportergenet kunne gro. Disse transformanter indeholder cdna, der koder for protein med affinitet for vores DNA element. 22
24 To-hybrid teknikker Ofte er man interesseret i at undersøge, om to proteiner eller proteinfragmenter interagerer in vivo. Ud over immunoprecipitation efterfulgt af western blotting er tohybridteknikken den eneste mulighed. To-hybridteknikken udnytter, at transkriptionsfaktorer er opbyggede af mindst to uafhængige domæner; det DNA bindende domæne og det transkriptionsaktiverende domæne. Det DNA bindende domæne er i stand til at genkende en bestemt sekvens i en promotor/enhancer region, medens det transkriptionsaktiverende domæne er i stand til at interagere med det generelle transkriptionsapparat og aktivere transkription. Hvis vi vil vise at to proteiner X og Y interagerer, kan vi fremstille et plasmid der koder for en in frame fusion mellem det DNA bindende domæne fra gærens GAL4 protein (GAL4 DNA )og protein X. Expression af dette fusionsprotein vil bevirke, at der dannes et protein, der vil kunne binde til DNA indeholdende GAL4 proteinets genkendelsessekvens. Samtidigt fremstiller vi et plasmid, der koder for en in frame fusion mellem det transkriptionsaktiverende domæne fra GAL4 (GAL4 TR ) og protein Y. Hvis protein X og protein Y kan interagere, vil de danne et proteinkompleks, hvorved det transkriptionsaktiverende domæne bliver bundet til en promotor med GAL4 genkendelsessekvens og transkriptionen vil aktiveres. Indeholder værstorganismen en transkriptionel fusion mellem en promotor, hvis aktivitet er afhængig af GAL4, og et reportergen, vil dette kun blive udtrykt hvis protein X og protein Y interagerer stabilt. Reportergenet kan være en autotrof markør (f.eks. et LYS2 gen) eller lacz fra E. coli. Aktivering af reportergenet vil således gøre en lys2 stamme autotrof (den kan leve uden lysin i mediet) eller gøre stammen blå på en X-gal plade. Systemet kan også bruges til at fiske cdna kloner, der koder for protein, som er i stand til at binde et bestemt kendt protein. Ønsker vi f.eks. at bestemme, hvilke proteiner der interagerer med protein X, vil vi fremstille et cdna bibliotek i en vektor der indeholder GAL4 TR. Biblioteket vil vi fremstille så vi opnår in frame fusioner mellem GAL4 TR og vores indsatte cdna. Kun cdna kloner, der koder for et protein der binder til X, vil give anledning til transkription af reportergenet. Er reportergenet et autotrof markør vil de rigtige gærkolonier kunne selekteres på plader. Her har vi beskrevet to-hybridsystemet i gær under anvendelse af GAL4 proteinet. Der findes forskellige variationer over dette tema, så som anvendelse af andre DNA bindende proteiner og transkriptionsaktiverende domæner. Endvidere er der udviklet to-hybridsystem til anvendelse i E. coli. Teknikken er illustreret i Figur 17 23
25 GAL4 DNA X P DNA GAL4 -X Y GAL4 TR TR P GAL -Y GAL4 TR X Y GAL4 DNA GAL4 UAS Reportergen Figur 17: Illustration af to-hybrid teknikken: En gærstamme transformeres med to plasmider, der udtrykker GAL4 DNA -X fusionen henholdsvis GAL TR -Y fusionen fra en konstitutive promotorer (P). Gærstammen indeholder på sit kromosom en fusion mellem minimalpromotor (TATA box) og GAL4 UAS (upstream activating sequence), som er nødvendig for effektiv transkription fra promotoren. Hvis protein X binder til protein Y, bliver det transkriptionsaktiverende domæne bragt i position til at kunne aktivere transkription, og reportergenet bliver transkriberet. I litteraturen kaldes X ofte for bait og Y for prey. 24
26 Bestemmelse af polysomfordelingen i eukaryote celler Ofte er det interessant at vide, hvorledes frekvensen af translationsinitieringer reagerer på en bestemt fysiologisk påvirkning. Da translationen normalt er reguleret på initieringsniveau, vil antallet af ribosomer på det enkelte mrna molekyle afspejle initieringsfrekvensen. For at fastfryse ribosomerne på mrnaet under oprensning tilsættes cykloheximid, som inhiberer elongering af ribosomerne. Herefter kan cellerne lyseres og celleekstraktet påsættes en sukrosegradient, der separerer efter densitet. Sædvanligvis anvender man en gradient der indeholder fra 15-47% sukrose. Jo flere ribosomer, der sidder på mrnaet, jo længere migrerer ribosom-mrna komplekset i sukrosegradienten. Efter ultracentrigering pumpes gradienten ud af centrifugerøret og ml fraktioner opsamles. På vej til fraktionsopsamleren passerer væsken en UV-recorder, der måler UV absorptionen ved 260 nm. Ved denne bølgelængde er absorptionen propertional med indholdet af nukleinsyre. Figur 18 viser i skematisk form forsøgsopstillingen. Figur 18 viser udprintet fra computeren der kontinuert opsamler UV data. UV absorptionen er angivet som funktion af det volumen, der er pumpet ud af gradienten. Den viste UV profil er karakteristisk for en eukaryot celle med aktiv proteinsyntese. Ønsker man at bestemme, hvor i gradienten et givet mrna molekyle befinder sig (og dermed om det er i polysom, monosom eller nøgent RNA fraktionen) kan det gøres ved Northern blotting, RNase protection eller real-time PCR på de opsamlede fraktioner. 25
27 Computeropsamling af data UV monitor 15% 47% Pumpe 47% sukrose Fraktionsopsamler Figur 18: Skematisk oversigt over instrumentparken til bestemmelse af polysomfordelingen i eukaryote celler. Et typisk opsamlet datasæt er vist i Figur
28 Polysomer Figur 19: Resultatet af en polysomanalyse fra en levercelle med aktiv proteinsyntese. Positionen af 40S, 60S, monosomer (M) og polysomer er angivet. Sukrosekoncentrationen stiger fra højre mod venstre. 27
29 Filterbindingsassay En alternativ metode til undersøgelse af binding mellem protein of DNA ved EMSA er filterbindingsassay. Metoden bygger på det princip at nitrocellulosemembraner ikke binder dobbelstrenget DNA men protein. Dobbelstrenget DNA tilbageholdes derfor kun af filteret, hvis det er bundet til et protein. Dette simple assay kan anvendes til at kvantificere protein-dna interaktionen. Dette kan også bruges til at separarere enkelstrenget DNA fra dobbeltstrenget. Som i et EMSA assay anvendes radioaktivt mærkede dobbelstrengede oligonukleotider. Princippet i assayet er vist i Figur
30 A B C Figur 20: Princippet i filterbindingsassay. Dobbelstrenget DNA løber igennem nitrocellulose membranen, medens protein tilbageholdes. DNA, der er bundet til protein, tilbageholdes derfor også. Anvendes radioaktivt mærket DNA kan den tilbageholdte mængde DNA bestemmes ved scientillationstælling. 29
31 Site-directed mutagenese ved overlap extension Ofte ønsker man at ændre på nukleotidsekvensen i et klonet stykke DNA. Det kan være for at undersøge betydningen af enkelte nukleotider for binding af en transkriptionsfaktor eller for at ændre på den primære struktur af et protein. Den nemmeste måde at gøre det på, er ved hjælp af PCR. Figur 21 illustrerer teknikken og Figur 22 giver et konkret eksempel. Figur 21: Skematisk oversigt over den af Ho et al (1989) beskrevne site-directed mutagenese protokol. Dobbelstrenget DNA templates og syntetiske DNA primere er angivet som orienterede linjer. Pilen angiver 5'- 3' orienteringen af den enkelte DNA streng. Mutationsstedet er angivet med en sort firkant. PCR primere er angivet med små bogstaver mens PCR produkter er angivet med store bogstaver, som angiver hvilke primere, der blev brugt til PCR amplifikationen. Den indrammede del af figuren angiver intermediæren under den afsluttende PCR reaktion, hvor de denaturerede initiale PCR fragmenter anneales i de overlappende regioner og hvor de frie 3' ender forlænges af DNA polymerasen. Resultatet er et PCR fragment indeholdende den ønskede mutation. Dette DNA fragment kan nu klones tilbage i plasmidet, idet PCR fragmentet er planlagt således at det i enderne indeholder genkendelsesekvenser for restriktions enzymer. 30
32 For at konkretisere metoden fra Figur 20 viser nedenstående figur sekvenserne af de primere der skal bruges for at ændre den med fed skrift angivne adenin til cytosin. 5 GTCATAGCGGATTAGCAAAG GCTTACGTAAGCTGACTGGC CAGTATCGCCTAATCGTTTC CGAATGCATTCGACTGACCG TAGCTAGCCGGATTTCGATGCTG ATCGATCGGCCTAAAGCTACGAC 5 C PCR 1: PCR fragment genereret med primeren 5 GTCATAGCGGATTAGCAAAG 3 Og mutant-primeren 5 GCCAGTCAGCGTACGTAAGC 3. Dette PCR fragment har sekvensen 5 GTCATAGCGGATTAGCAAAG GCTTACGTACGCTGACTGGC 3 3 CAGTATCGCCTAATCGTTTC CGAATGCATGCGACTGACCG 5 PCR 2: PCR fragment genereret med primeren 5 CAGCATCGAAATCCGGCTA 3 og Mutant-primeren 5 GCTTACGTACGCTGACTGGC 3. Dette PCR fragment har sekvensen 5 GCTTACGTACGCTGACTGGC TAGCTAGCCGGATTTCGATGCTG 3 3 CGAATGCATGCGACTGACCG ATCGATCGGCCTAAAGCTACGAC 5 PCR 3: PCR fragment generet med fragmenterne fra de to initielle PCR reaktioner som template og primerne 5 GTCATAGCGGATTAGCAAAG 3 og 5 CAGCATCGAAATCCGGCTA 3 (de to yderprimere ). Dette fragment har sekvensen 5 GTCATAGCGGATTAGCAAAG GCTTACGTACGCTGACTGGC CAGTATCGCCTAATCGTTTC CGAATGCATGCGACTGACCG TAGCTAGCCGGATTTCGATGCTG ATCGATCGGCCTAAAGCTACGAC 5 Figur 22: Et konkret tilfælde for at illustrere metoden der er skematiseret i Figur
33 Alternativ site directed mutagenese protokol En anden metode til site directed mutagenese er at in vitro replikere et plasmid ved hjælp af en primer indeholdede de(n) ønskede nukleotidsubstitutioner og en termostabil DNA polymerase. Princippet i metoden er vist nedenfor og bygger på at mutantprimeren indbygges i det nye plasmid som resultat af DNA syntesen. Samtidig gøres der brug af restriktionsenzymet DpnI, som kun skærer sekvenser som er enten fuldt metylerede eller hemimetylerede. Skæres DNAet efter endt syntese vil de oprindelige template strenge blive nedbrudt og dermed ikke kunne transformere E. coli. Da syntesen af DNA sker i tilstedeværelse af u-methylerede nukleotider vil de nysyntetiserede plasmider ikke blive skåret, og derfor give anledning til transformanter i E. coli. Derved opnår man en forøget frekvens af plasmider indeholdende de(n) ønskede substitutioner. 32
34 Figur 23 : Alternativ site-directed mutagenses protokol til overlap extension metoden. De to strenge i template DNAet er vist som rød henholdsvis blå. Mutantprimerne er angivet som pile. Et gult kryds angiver inkorporering af nukleotidsubstitutionen. Efter den første replikationsrunde vil der være plasmider med en mutant streng og en vildtype streng. Efter den anden replikationsrunde vil der være 2 plasmider med mutation i begge strenge og to plasmider med en muteret streng og en vildtype streng, Jo flere replikationsrunder jo flere korrekte plasmider. Hvis nogle plasmider slet ikke replikeres, vil de selvfølgelig også være tilstede. DpnI skæring fjerner plasmider med to eller en vildtype streng. 33
35 Fusionering af to DNA fragmenter ved hjælp af PCR. Den i Figur 21 viste metode til site-directed-mutagenese kan også bruges til præcist at fusionere to DNA fragmenter. Princippet bygger igen på overlap extension. Ønsker vi at fusionere de to nedenstående DNA fragmenter på de viste steder kan vi generere to initielle PCR fragmenter, der har sekvensidentitet der, hvor den endelige fusionering skal foregå. PCR 1: PCR på den øverste template med primerne A og B. PCR 2: PCR på den øverste template med primerne C og D. PCR 3: De to initielle PCR produkter blandes sammen og der foretages PCR med primer A og D. PCR 1 PCR 2 PCR 3 34
36 Proteomics. De analyser, det er blevet muligt at udføre på hele cellers proteinsammensætning, går under fællesbetegnelsen proteomics. Proteomet er således en beskrivelse af hele cellens indhold af protein; hvilke der er tilstede under bestemte fysiologiske omstændigheder og hvor meget der er af det enkelte protein. Oftest udføres analysen ved at koble to-dimensionel gelelektroforese med massespektrometri. Der kan dog også anvendes søjlekromatografiske metoder til at adskille proteomet før identifikationen ved massespektrometri. To-dimensionel gelektroforese udføres ved, at et proteinekstrakt separeres ved isoelektriosk fokusering i en immobiliseret ph gradient. Under den isoelektriske fokusering vandrer det enkelte protein i et elektrisk felt, indtil det når et område med en ph værdi hvor proteinets nettoladning er nul. Herefter er det upåvirkede af det elektriske felt og forbliver på denne position. Separering i den første dimension sker i en en lille strip som er nogle få millimeter tyk og typisk 18 eller 24 cm lang. Efter denne separation ligger proteiner adskilt som i en stregkode fra Brugsen. Da mange proteiner har det samme isoelektriske punkt, er der flere proteiner tilstede i hver streg i stregkoden. Resultatet ses i Figur 24. For at adskille proteiner med det samme isoleketriske punkt foretages en adskillelse efter størrelse. Dette gøres ved at separere proteinerne ved SDS-PAGE (poly-akryl-amid-gelelektroforese i tilstedeværelse af detergentet sodium-dodecylsulphate). Denne adskillelse sker efter størrelse. Rent praktisk foregår det ved, at strippen lægges ovenpå SDS-PAGE gelen og fæstnes med agarose. Derefter pålægges gelen et elektrisk felt og de SDS-bundne proteiner vandrer med en mobilitet, der er afhængig af deres størrelse. Efter endt kørsel visualiseres proteinerne typisk ved sølvfarving, som kan detektere proteiner i ng området. Resultatet ses i figur 24. En sådan 2-dimensionel gel kan separere op mode 2000 proteiner. Da en typisk eukaryot celle udtrykker væsentlig flere proteiner, kan følsomheden øges ved at anvende 1. dimensions strips, der separerer proteiner i mindre ph intervaller. Man kan således få strips der separerer fra ph 3 5.6, , , 7 11 eller ph 3 til ph 11. Formålet med den to-dimensionelle separation af protein er oftest at bestemme ændringer i cellens proteom som resultat af en fysiologisk ændring. Det er derfor essentielt at kunne bestemme identiteten af de proteiner hvis koncentration ændres. Da vi ønsker beskrivelse af hele proteomet der det derfor vigtigt, at identifikation kan foregå hurtigt og nemt. Dette er i dag muligt på grund af udviklingen indenfor to områder; sekventering og bioinformatisk analyse af hele genomer samt massespektrometri. Går man ind på nettet på adressen kan man se hvor mange genomer der er sekvensbestemt. Ud fra en genomsekvens er det muligt at komme med kvalificerede gæt på, den primære struktur af hver enkelt protein den enkelte organisme kan producere. Med den primære struktur til rådighed kan man forudsige, hvorledes et sekvensspecifik proteolytisk enzym vil spalte hver enkelt protein. Et sådant enzym er serin proteasen trypsin, som spalter efter lysin og arginin. Vi kan således beregne, hvor store peptidfragmenter trypsinbehandling af hvert enkelt protein i cellen vil generere. Nu er det så heldigt, at trypsinfragmenterne udgør et fingeraftryk af hvert enkelt protein. Kendskab til de tryptiske fragmenters størrelse kan således for mere end 75% af alle proteiners vedkommende bruges til at identificere proteinet. Her kommer massespektrometri ind i billedet. Massespektrometri er en ekstrem nøjaktik 35
37 teknik til bestemmelse af proteiners og peptiders molekylvægt (se lærebogen afsnit 24.4 Proteomics til side 845 nederst). Med kendskab til molekylvægten af de tryptiske fragmenter og den organisme proteinet er isoleret fra kan vi gå på internettet på adressen og få at vide, hvilket protein vi har isoleret fra vores 2-D gel. 1. Dimension. 2. Dimension Figur 23: Illustration af hvorledes proteinerne er fordelt efter isoelektrisk fokusering i den 1. dimension og efter separation i den 2. dimension. Efter 2. dimension fremstår proteinerne som pletter, hvor størrelsen og intensiteten i pletten siger noget om proteinmængden. I en rigtig gel vil der være om mod et par tusinde pletter, som kan identificeres. De interessante pletter skæres ud af gelen og proteinerne fordøjes i gelen med trypsin, hvorefter de tryptiske fragmenter oprenses og deres molekylvægt bestemmes ved massespektrometri. 36
38 Transkriptomanalyse Hvor 2-D elektroforese giver information om proteomets sammesætning, er der ved hjælp af microarray teknologi mulighed for at bestemme transkriptomets sammensætning. Transkriptomet udgøres af samtlige mrna molekyler i cellen. D.v.s. at en transkriptomanalyse giver information om hvilke mrna der findes i en givet celle på et givet tidspunkt samt hvad koncentrationen af det enkelte mrna molekyle er. I det følgende vil vi kigge på den teknologi, der anvendes af firmaet Affymetrix ( Som det var tilfældet med de tidligere beskrevne mrna analyser (northern blotting, RNase protektion og real time PCR) bygger microa array teknologien på at bestemte nukleinsyresekvenser kan identificeres ved hjælp et oligonukleotid med komplementær sekvens og mængden af den bestemte nukleinsyresekvens kan kvantifiseres.forudsætningen for at kunne fremstille en microarray chip er at nukleotidsekvensen af hele organismen er kendt og at nukleotidsekvensen af alle de mrna molekyler organismen kan producere kan estimeres ved en bioinformatisk analyse. Er dette tilfældet kan man designe oligonukleotider (prober) der vil være specifikke for hvert enkelt transkript.hver probe er 25 nukleotider lang og syntetiseres direkte på en glasoverflade med en fotolitografisk proces udviklet til elektronikindustrien. For at øge nøjaktigheden i kvantifikationen syntetiseres der 22 forskellige nukleotider til genkendelse af hvert transkript. Hver probe hybridiserer så til hver sit område af det enkelte mrna molekyle.da det er vigtigt at kunne estimere den uspecifikke binding mellem den enkelte probe og det enkelt mrna molekyle, syntetiseres der også for hver probe en mis-match probe (en probe indeholdende en nukleotidsubstitution i midten af proben, hvilket under optimale hybridiseringsomstændigheder vil forhindre binding til den korrekte target sekvens).metoden til syntese af de mange prober på glaspladen er vist i Figur 25. For at kunne kvantificere den mængde mrna der er hybridiseret til hver enkelt probe er det nødvendig med en detektionsmetode der er simpel og meget følsom. Derfor mærkes RNAet før det hybridiseres til micro array chippen med biotin, som flourescerer efter bestråling med laserlys. D.v.s. at laseren exciterer den inkorporerede biotin og måler flourescence fra hvert lille område af chippen hvor der sidder oligonukleotider. Da der sidder > prober per cm 2 er det vigtigt, at der kun måles fluorescens fra et sæt prober adgangen. 37
39 Figur 25: Metode til syntese af prober direkte på glaspladen. Som nævnt sker der syntese i > punkter per cm 2. Koncepter er, at der fremstilles en lang række net, der kun tillader lys at slippe igennem de steder, hvor der skal kobles et nyt nukleotid på. Der skal således skiftes net efter hvert syntesetrin. Protokollen for mærkning af mrnaet med biotin og hybridisering til microaary chippen er vist i Figur
40 Figur 26: Protol til mærkning af mrna og hybridisering til micro array chippen. 39
41 Differential display Konceptet bag denne teknik er at visualisere alle mrna molekylær i en celle under to forskellige fysiologiske omstændigheder for at identificere hvilke mrna molekyler, der er differentielt udtrykt Teknikken giver et sekvensen af et stykke cdna der repræsenterer et differentielt udtrykt gen. Sekvensen af den isolerede cdna stump bruges efterfølgende til at klone fuldlængde cdna. Som vist i Figur 27 kan mrnadeles op i fire grupper efter hvilket nukleotid, der sidder før det første A i polya halen 5 A AAAAAAA 3 5 G AAAAAAA 3 5 C AAAAAAA 3 5 T AAAAAAA 3 Figur 27: Strukturen af de tre klasser cellens mrna kan inddeles i. Revers transkription med polydtc, polydtg eller polydta primer vil generere cdna fra ca. 1/3 af cellens mrna (Hvis der er et eller flere A er før polya halen kommer der på et tidspunkt et G, C eller T). Klassen af mrna med et A før polya halen vil såles kunne underopdeles i tre klasser hvor nukleotidet før det første A er enten G,C, eller T. For at generere tilfældige fragmenter fra forskellige mrna sekevnser er det nødvendigt at amplificere sit revers transkriberede DNA med en primer som annealer til en sekvens der er så kort, at den findes i de fleste mrna molekyler. Dette sikres ved at anvende en relativ kort primer på 13 nukleotider. Dog kan vi ikke bruge samtlige 13 merer da vi så ville få alt for mange PCR produkter til at vi praktisk kan håndtere dem. Samtidigt er det vigtigt, at revers transkriptions primeren altid binder det samme sted til 3 enden af mrnaet. Det sikres ved, at have enten et G, C eller T i denne primers 3 ende. Primeren kan da kun fungere, hvis den binder korrekt til polya halen. Figur 28 viser hvorledes der genereres DNA fragmenter ved PCR amplifikationen med polydt primeren og de tilfældige 13 merer. 40
42 3 OH 5 p cdna mrna CTTTTTTTTT 5 p GAAAAAAAA 3 OH Tilsæt en blanding af 40 tilfældige 13 nukleotider lange primere ( ) og polydtc primeren ( ) 3 OH 3 OH 3 OH 3 OH 3 OH 3 OH 3 OH 3 OH 3 OH 3 OH CTTTTTTTTT 5 p CTTTTTTTTT 5 p CTTTTTTTTT 5 p CTTTTTTTTT 5 p CTTTTTTTTT 5 p CTTTTTTTTT 5 p CTTTTTTTTT 5 p CTTTTTTTTT 5 p CTTTTTTTTT 5 p CTTTTTTTTT 5 p PCR α [ 32 P]-GTP Figur 28: Skematisk oversigt over hvorledes den faste position af revers transkriptionsprimeren og bindingsstedet i det enkelte mrna molekyle giver PCR fragmenter af forskellig længde fra forskellige mrna molekyler. For at øge sensitiviteten i assayet tilsættes α-[ 32 P]-GTP til reaktionen For at visualisere alle de genererede cdna fragmenter separeres de i en denaturerende poly-akrylamid-gel (indeholder urinstof) De separerede bånd visualiseres ved PhosphoImage analyse eller autoradiografy. Figur 29 viser i skematisk form resultatet af et differential display forsøg hvor en kontrol celle er sammenlignet med den den rigtige prøve. F.eks. en normal celle og en cancercelle. 41
43 Kontrol Prøve Differentielt udtrykt mrna Figur 29: resultatet af et differential display experiment, hvor mrna mønsteret sammenlignes mellem en kontrol celle og den rigtige celleprøce. Bånd der forsvinder eller opstår i prøven er interessante og repræsenterer differentialt udtrykte gener. cdna, der repræsenterer differentielt udtrykt mrna, kan ekstraheres fra gelen og PCR amplificeres med den anvendte polydt primeren og de 40 tilfældige 13 merer. Dernæst sekventeres PCR fragmentet. Sekvensen kan bruges til at screene et cdna bibliotek eller til at designe primere til 5 og 3 RACE 42
44 Ekspressionskloning i Xenopus oocytter Når det eneste håndtag, man har for at screene et cdna bibliotek, er den aktivitet, det tilhørende protein besidder, er ekspressionskloning den eneste mulighed. Ekspressionskloning af cdna kodende for membranproteiner tager udgangspunkt i æg (oocytter) fra den afrikanske klofrø, Xenopus laevis. Oocytter herfra har den fordel, at de e 3-5 mm store, hvilket tillader injektion i oocytter med nogle få nl RNA eller DNA opløsning via en meget tynd glaspipette. Oocytten har en høj kapacitet for proteinsyntese så det injicerede RNA vil blive translateret effektivt og proteinet vil blive transporteret til den rigtige membran. Man kan injicere RNA oprenset fra væv eller man kan injicere in vitro fremstillet RNA. Oocytternes store diameter gør også, at man kan anvende dem til elektrofysiologiske eksperimenter. Dette er relevant, hvis man udtrykker en ion-kanal, idet transport gennem kanalen vil forårsage, at der løber en strøm gennem oocyttens membran. Denne strøm kan så måles. Så vil vi gerne undersøge, om der findes en eller flere K-kanaler i hjernevæv, kan vi oprense polya RNA fra hjerne og injicere dette i oocytten. Kan vi nu måle en K + afhængig strøm gennem oocytten, må vi konkluderem at hjernevævet indeholder polya RNA, kodende for en K-kanal. Næste trin vil være, at vi gerne vil klone cdna kodende for de K-kanaler, der findes i vores hjernevæv. Her er problemet, at vi intet kender til kanalen, bortset fra at de kan transportere K + ioner. Kanaler forekommer i så lille koncentration i nativt væv, at det umuliggør oprensning og sekventering af proteinet. Anvendelse af en probe er derfor udelukket. K-kanal mrna udgør måske 0.05% af det total mrna, så vi er virkelig ude at lede efter en nål i en meget stor høstak. For at komme igang, vil vi først størrelsesfraktionere vores polya RNA fra hjerne for at finde de(n) længder af mrna, der koder for K-kanalen/K-kanalerne. Dette kan man gøre ved ultracentrifugering i en sukrosegradient, som omtalt under afsnittet polysom analyse. Vi udtager så fraktioner fra gradienten og injicerer dem en af gangen i oocytterne og måler elektrofysiologisk, om der går en K + strøm gennen oocymembranen. Den fraktion, der giver positivt signal, anvender vi til at fremstille et cdna bibliotek i en vektor, der tillader in vitro transkription. Biblioteket, der typisk vil indeholde kolonier, plades ud på 10 petriskåle. Hvis andelen af K-kanal mrna er 0.05 % vil hver petriskål, der indeholder 4000 kolonier indeholde 2 rigtige kolonier. 43
Besvarelse af opgaverne til den Spm.A: efter TGA TCA Spm. B:
 Besvarelse af opgaverne til den 20-9-06 Spm.A: Her vil vi gerne sætte et relativt lille stykke DNA (FLAG) sammen med et relativt stort stykke (PRB1). Det lille stykke er for lille til at vi kan PCR amplificere
Besvarelse af opgaverne til den 20-9-06 Spm.A: Her vil vi gerne sætte et relativt lille stykke DNA (FLAG) sammen med et relativt stort stykke (PRB1). Det lille stykke er for lille til at vi kan PCR amplificere
Løsninger til opgaverne den Spm. A: Spm. B Spm. C:
 Løsninger til opgaverne den 12-9-07 Spm. A: Her er vi i en situation at det eneste der kendes til vores receptor er, at den kan binde en ligand, som vi har til rådighed i en radioaktiv mærket version.
Løsninger til opgaverne den 12-9-07 Spm. A: Her er vi i en situation at det eneste der kendes til vores receptor er, at den kan binde en ligand, som vi har til rådighed i en radioaktiv mærket version.
Spm. A: Hvad viser data i Figur 1?
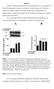 Opgave 1 Leptin er et plasma proteinhormon der primært produceres af og secerneres fra fedtceller (adipocytter). Leptin, der er kodet af ob (obecity) genet, er 16 kda stort. Leptins fysiologiske funktion
Opgave 1 Leptin er et plasma proteinhormon der primært produceres af og secerneres fra fedtceller (adipocytter). Leptin, der er kodet af ob (obecity) genet, er 16 kda stort. Leptins fysiologiske funktion
En forsker har lavet et cdna insert vha PCR og har anvendt det følgende primer sæt, som producerer hele den åbne læseramme af cdna et:
 F2011-Opgave 1. En forsker har lavet et cdna insert vha PCR og har anvendt det følgende primer sæt, som producerer hele den åbne læseramme af cdna et: Forward primer: 5 CC ATG GGT ATG AAG CTT TGC AGC CTT
F2011-Opgave 1. En forsker har lavet et cdna insert vha PCR og har anvendt det følgende primer sæt, som producerer hele den åbne læseramme af cdna et: Forward primer: 5 CC ATG GGT ATG AAG CTT TGC AGC CTT
Løsninger til opgaverne den Opgave 2 januar 2003 Spm. 1:
 Løsninger til opgaverne den 6-9-06 Opgave 2 januar 2003 Spm. 1: Fidusen er her at vi kender den primære struktur af glukoamylasen fra tre forskellige svampe, en ascomycet, en basidiomycet og en zygomycet.
Løsninger til opgaverne den 6-9-06 Opgave 2 januar 2003 Spm. 1: Fidusen er her at vi kender den primære struktur af glukoamylasen fra tre forskellige svampe, en ascomycet, en basidiomycet og en zygomycet.
CYP7A1 (0,1 nm) CYP7A1 (0nM) UBIC (0,1 nm)
 Opgave 1 Leveren spiller en central rolle i lipid metabolismen og i opretholdelse af lipid homeostasen i hele kroppen. Lipidmetabolismen er fejlreguleret hos blandt andet svært overvægtige personer samt
Opgave 1 Leveren spiller en central rolle i lipid metabolismen og i opretholdelse af lipid homeostasen i hele kroppen. Lipidmetabolismen er fejlreguleret hos blandt andet svært overvægtige personer samt
datp, dctp, dgtp, dttp, [α 32 P]-dCTP urinstofholdig polyacrylamid gel røntgen film
![datp, dctp, dgtp, dttp, [α 32 P]-dCTP urinstofholdig polyacrylamid gel røntgen film datp, dctp, dgtp, dttp, [α 32 P]-dCTP urinstofholdig polyacrylamid gel røntgen film](/thumbs/52/30168242.jpg) Opgave 1 Denne opgave omhandler osteoblast differentiering. Osteoblast celler er involveret i opbygning af knoglevæv. Osteoblast celler dannes ud fra såkaldte calvarial celler. For at forstå det molekylære
Opgave 1 Denne opgave omhandler osteoblast differentiering. Osteoblast celler er involveret i opbygning af knoglevæv. Osteoblast celler dannes ud fra såkaldte calvarial celler. For at forstå det molekylære
PCR (Polymerase Chain Reaction): Opkopiering af DNA
 PCR (Polymerase Chain Reaction): Opkopiering af DNA PCR til at opkopiere bestemte DNA-sekvenser i en prøve er nu en af genteknologiens absolut vigtigste værktøjer. Peter Rugbjerg, Biotech Academy PCR (Polymerase
PCR (Polymerase Chain Reaction): Opkopiering af DNA PCR til at opkopiere bestemte DNA-sekvenser i en prøve er nu en af genteknologiens absolut vigtigste værktøjer. Peter Rugbjerg, Biotech Academy PCR (Polymerase
Besvarelse til opgave 1 januar 1999 Spm. A: Spm. B:
 Besvarelse til opgave 1 januar 1999 Spm. A: Vi må lave et genomisk bibliotek i en lambdafag, cosmid, BAC eller YAC plasmid. Til dette vil vi skære det genomiske DNA partielt med Sau3A, så vi får stykker
Besvarelse til opgave 1 januar 1999 Spm. A: Vi må lave et genomisk bibliotek i en lambdafag, cosmid, BAC eller YAC plasmid. Til dette vil vi skære det genomiske DNA partielt med Sau3A, så vi får stykker
Udarbejdet af Anna-Alexandra Grube Hoppe
 METODER - BIOKEMI For medicinstuderende - Panuminstituttet UDARBEJDET AF ANNA-ALEXANDRA GRUBE HOPPE RESTRIKTIONSENZYM ANALYSE (E: 329-330) Restriktionsenzym analyse er en metode, der bruges til at kortlægge
METODER - BIOKEMI For medicinstuderende - Panuminstituttet UDARBEJDET AF ANNA-ALEXANDRA GRUBE HOPPE RESTRIKTIONSENZYM ANALYSE (E: 329-330) Restriktionsenzym analyse er en metode, der bruges til at kortlægge
Figur 1: Strukturelt kort over plasmiderne pyeast og pmamal anvendt til undersøgelse af
 Opgave 2 juni 1999 DNA rekombination er vigtig for eukaryote celler i forbindelse med meiosen. Samtidig er rekombination mellem fremmed DNA og endogen DNA essentiel i fremstillingen af transgene organismer
Opgave 2 juni 1999 DNA rekombination er vigtig for eukaryote celler i forbindelse med meiosen. Samtidig er rekombination mellem fremmed DNA og endogen DNA essentiel i fremstillingen af transgene organismer
DNA origami øvelse 2013. DNA origami øvelse
 DNA origami øvelse Introduktion I denne øvelse bruger vi DNA origami teknikken til at samle en tavle af DNA med dimensioner på 70 nm x 100 nm. Tavlen dannes af et langt enkeltstrenget DNA molekyle, der
DNA origami øvelse Introduktion I denne øvelse bruger vi DNA origami teknikken til at samle en tavle af DNA med dimensioner på 70 nm x 100 nm. Tavlen dannes af et langt enkeltstrenget DNA molekyle, der
Velkommen. Test dit eget DNA med PCR. Undervisningsdag på DTU Systembiologi. Undervisere:
 Velkommen Test dit eget DNA med PCR Undervisningsdag på DTU Systembiologi Undervisere: Hvem er I? 2 DTU Systembiologi, Danmarks Tekniske Universitet Hvilke baser indgår i DNA? A. Adenin, Guanin, Cytosin,
Velkommen Test dit eget DNA med PCR Undervisningsdag på DTU Systembiologi Undervisere: Hvem er I? 2 DTU Systembiologi, Danmarks Tekniske Universitet Hvilke baser indgår i DNA? A. Adenin, Guanin, Cytosin,
Restriktionsenzymer findes Genkendelsessekvens
 Restriktionsenzymer Skanning Restriktionsenzymer findes i bakterier (forsvarsmekanisme) IKKE i eukaryote celler 3-5 - 5-3 - Restriktionsenzyme Genkendelsessekvens Palindromisk Otto, Anna, radar Madam I
Restriktionsenzymer Skanning Restriktionsenzymer findes i bakterier (forsvarsmekanisme) IKKE i eukaryote celler 3-5 - 5-3 - Restriktionsenzyme Genkendelsessekvens Palindromisk Otto, Anna, radar Madam I
Studienummer: MeDIS Exam 2015. Husk at opgive studienummer ikke navn og cpr.nr. på alle ark, der skal medtages i bedømmelsen
 MeDIS Exam 2015 Titel på kursus: Uddannelse: Semester: Videregående biokemi og medicinudvikling Bachelor i Medis 5. semester Eksamensdato: 26-01-2015 Tid: kl. 09.00-11.00 Bedømmelsesform 7-trin Vigtige
MeDIS Exam 2015 Titel på kursus: Uddannelse: Semester: Videregående biokemi og medicinudvikling Bachelor i Medis 5. semester Eksamensdato: 26-01-2015 Tid: kl. 09.00-11.00 Bedømmelsesform 7-trin Vigtige
DNA origami øvelse. Introduktion. DNA origami øvelse 3 timer 2018
 DNA origami øvelse Introduktion I denne øvelse bruger vi DNA origami teknikken til at samle en tavle af DNA med dimensioner på 70 nm x 100 nm. Tavlen dannes af et langt enkeltstrenget DNA molekyle, der
DNA origami øvelse Introduktion I denne øvelse bruger vi DNA origami teknikken til at samle en tavle af DNA med dimensioner på 70 nm x 100 nm. Tavlen dannes af et langt enkeltstrenget DNA molekyle, der
DNA origami øvelse DNA origami øvelse
 DNA origami øvelse Introduktion I denne øvelse bruger vi DNA origami teknikken til at samle en tavle af DNA med dimensioner på 70 nm x 100 nm. Tavlen dannes af et langt enkeltstrenget DNA molekyle, der
DNA origami øvelse Introduktion I denne øvelse bruger vi DNA origami teknikken til at samle en tavle af DNA med dimensioner på 70 nm x 100 nm. Tavlen dannes af et langt enkeltstrenget DNA molekyle, der
DNA origami øvelse DNA origami øvelse
 DNA origami øvelse Introduktion I denne øvelse bruger vi DNA origami teknikken til at samle en tavle af DNA med dimensioner på 70 nm x 100 nm. Tavlen dannes af et langt enkeltstrenget DNA molekyle, der
DNA origami øvelse Introduktion I denne øvelse bruger vi DNA origami teknikken til at samle en tavle af DNA med dimensioner på 70 nm x 100 nm. Tavlen dannes af et langt enkeltstrenget DNA molekyle, der
Velkommen. Test dit eget DNA med PCR. Undervisningsdag på DTU Systembiologi. Undervisere: Sebastian, Louise og Ana
 Velkommen Test dit eget DNA med PCR Undervisningsdag på DTU Systembiologi Undervisere: Sebastian, Louise og Ana Hvem er I? 2 DTU Systembiologi, Danmarks Tekniske Universitet Dagens program 9:00 10:00 Introduktion
Velkommen Test dit eget DNA med PCR Undervisningsdag på DTU Systembiologi Undervisere: Sebastian, Louise og Ana Hvem er I? 2 DTU Systembiologi, Danmarks Tekniske Universitet Dagens program 9:00 10:00 Introduktion
Spm. B: Hvad viser data i Figur 2?
 Opgave 1 Kernereceptorer (nuclear receptors) udgør en familie af ligandaktiverede transkriptionsfaktorer, der regulerer transkription af en række gener, som respons på tilstedeværelse af f.eks.lipofile
Opgave 1 Kernereceptorer (nuclear receptors) udgør en familie af ligandaktiverede transkriptionsfaktorer, der regulerer transkription af en række gener, som respons på tilstedeværelse af f.eks.lipofile
Opgave 1 Spm. A: Spm. B:
 Opgave 1 Familien af TM7 receptorer (receptorer med 7 transmembrane segmenter) udgør ud fra genomsekvensen den største proteinfamile i C. elegans. Det samme må forventes at gælde i pattedyr. Imidlertid
Opgave 1 Familien af TM7 receptorer (receptorer med 7 transmembrane segmenter) udgør ud fra genomsekvensen den største proteinfamile i C. elegans. Det samme må forventes at gælde i pattedyr. Imidlertid
GAPDH PCR modul Manual
 GAPDH PCR modul Manual Katalog nr. 166-5010EDU explorer.bio-rad.com Kopiering kun tilladt til undervisningsbrug Bemærk: Kittet indeholder temperaturfølsomme dele. Åbn derfor straks kassen og læg de pågældende
GAPDH PCR modul Manual Katalog nr. 166-5010EDU explorer.bio-rad.com Kopiering kun tilladt til undervisningsbrug Bemærk: Kittet indeholder temperaturfølsomme dele. Åbn derfor straks kassen og læg de pågældende
Menneskets væskefaser
 Menneskets væskefaser Mennesket består af ca. 60% væske (vand) Overordnet opdelt i to: Ekstracellulærvæske og intracellulærvæske Ekstracellulærvæske udgør ca. 1/3 Interstitielvæske: Væske der ligger mellem
Menneskets væskefaser Mennesket består af ca. 60% væske (vand) Overordnet opdelt i to: Ekstracellulærvæske og intracellulærvæske Ekstracellulærvæske udgør ca. 1/3 Interstitielvæske: Væske der ligger mellem
Formål At analysere for myosin i muskelvæv fra fisk ved brug af western blotting
 Center for Undervisningsmidler, afdeling København Western blotting Øvelsesvejledning Formål At analysere for myosin i muskelvæv fra fisk ved brug af western blotting Teori Proteomik er studiet af celleproteiners
Center for Undervisningsmidler, afdeling København Western blotting Øvelsesvejledning Formål At analysere for myosin i muskelvæv fra fisk ved brug af western blotting Teori Proteomik er studiet af celleproteiners
Dansk resumé for begyndere
 Dansk resumé for begyndere Dansk resumé for begyndere Dette afsnit introducerer bakteriel genregulation for enhver uden forudgående kendskab til dette emne. Alle nødvendige, videnskabelige betegnelser
Dansk resumé for begyndere Dansk resumé for begyndere Dette afsnit introducerer bakteriel genregulation for enhver uden forudgående kendskab til dette emne. Alle nødvendige, videnskabelige betegnelser
Test dit eget DNA med PCR
 Test dit eget DNA med PCR Navn: Forsøgsvejledning Formål med forsøget Formålet med dette forsøg er at undersøge jeres arvemateriale (DNA) for et transposon kaldet Alu. Et transposon er en DNA sekvens,
Test dit eget DNA med PCR Navn: Forsøgsvejledning Formål med forsøget Formålet med dette forsøg er at undersøge jeres arvemateriale (DNA) for et transposon kaldet Alu. Et transposon er en DNA sekvens,
Test dit eget DNA med PCR
 Test dit eget DNA med PCR Navn: Forsøgsvejledning Side 1 af 8 Formål med forsøget Formålet med dette forsøg er at undersøge jeres arvemateriale (DNA) for et transposon kaldet Alu. Et transposon er en DNA-sekvens,
Test dit eget DNA med PCR Navn: Forsøgsvejledning Side 1 af 8 Formål med forsøget Formålet med dette forsøg er at undersøge jeres arvemateriale (DNA) for et transposon kaldet Alu. Et transposon er en DNA-sekvens,
Test dit eget DNA med PCR
 Test dit eget DNA med PCR Forsøgsvejledning Navn: Side 1 af 7 Formål med forsøget Formålet med dette forsøg er at undersøge jeres arvemateriale (DNA) for et transposon kaldet Alu. Et transposon er en DNA
Test dit eget DNA med PCR Forsøgsvejledning Navn: Side 1 af 7 Formål med forsøget Formålet med dette forsøg er at undersøge jeres arvemateriale (DNA) for et transposon kaldet Alu. Et transposon er en DNA
Figur 1: A, nukleotidsekvensen af starten og slutningen af RPB1 genet. Initierings- og terminerings
 Opgave 2 juni 2004 Eukaryote celler indeholder 3 forskellige RNA polymeraser. Hver RNA polymerase er ansvarlig for transkription af bestemte klasser af gener. Ud fra mutationsanalyse i bagegær (Saccharomyces
Opgave 2 juni 2004 Eukaryote celler indeholder 3 forskellige RNA polymeraser. Hver RNA polymerase er ansvarlig for transkription af bestemte klasser af gener. Ud fra mutationsanalyse i bagegær (Saccharomyces
Test dit eget DNA med PCR
 Test dit eget DNA med PCR Navn: Forsøgsvejledning Side 1 af 8 Formål med forsøget Formålet med dette forsøg er at undersøge jeres arvemateriale (DNA) for et transposon kaldet Alu. Et transposon er en DNA
Test dit eget DNA med PCR Navn: Forsøgsvejledning Side 1 af 8 Formål med forsøget Formålet med dette forsøg er at undersøge jeres arvemateriale (DNA) for et transposon kaldet Alu. Et transposon er en DNA
Test dit eget DNA med PCR
 Test dit eget DNA med PCR Navn: Forsøgsvejledning Side 1 af 8 Formål med forsøget Formålet med dette forsøg er at undersøge jeres arvemateriale (DNA) for et transposon kaldet Alu. Et transposon er en DNA-sekvens,
Test dit eget DNA med PCR Navn: Forsøgsvejledning Side 1 af 8 Formål med forsøget Formålet med dette forsøg er at undersøge jeres arvemateriale (DNA) for et transposon kaldet Alu. Et transposon er en DNA-sekvens,
Biologi opgave Opsamling: Cellebiologi (Bioanalytiker modul3)
 1 Delphine Bonneau Biologi opgave Opsamling: Cellebiologi 1-6 Pelle har spist en kæmpe stor kage, og efterfølgende stiger hans blodsukker. Derfor sender kroppen besked til de endokrine kirtler i bugspytkirtlen
1 Delphine Bonneau Biologi opgave Opsamling: Cellebiologi 1-6 Pelle har spist en kæmpe stor kage, og efterfølgende stiger hans blodsukker. Derfor sender kroppen besked til de endokrine kirtler i bugspytkirtlen
Appendix 1: Udregning af mængde cellesuspention til udsåning. Faktor mellem total antal celler og antal celler der ønskes udsås:
 Appendix Appendix 1: Udregning af mængde cellesuspention til udsåning Fortyndingsfaktor: Efter trypsinering og centrifugering af celler fra cellestokken, opblandes cellerne i 1 ml medie. For at lave en
Appendix Appendix 1: Udregning af mængde cellesuspention til udsåning Fortyndingsfaktor: Efter trypsinering og centrifugering af celler fra cellestokken, opblandes cellerne i 1 ml medie. For at lave en
Proteiner. Proteiner er molekyler der er opbygget af "aminosyrer",nogle er sammensat af få aminosyrer medens andre er opbygget af mange tusinde
 Proteiner Proteiner er molekyler der er opbygget af "aminosyrer",nogle er sammensat af få aminosyrer medens andre er opbygget af mange tusinde Der findes ca. 20 aminosyrer i menneskets organisme. Nogle
Proteiner Proteiner er molekyler der er opbygget af "aminosyrer",nogle er sammensat af få aminosyrer medens andre er opbygget af mange tusinde Der findes ca. 20 aminosyrer i menneskets organisme. Nogle
Opgave 1 Slankemidler
 Opgave 1 Slankemidler Overvægt er et stigende problem i den vestlige verden, og der er derfor udviklet forskellige slankemidler. Et eksempel på et slankemiddel er Orlistat. Den kemiske struktur af Orlistat
Opgave 1 Slankemidler Overvægt er et stigende problem i den vestlige verden, og der er derfor udviklet forskellige slankemidler. Et eksempel på et slankemiddel er Orlistat. Den kemiske struktur af Orlistat
Analyse af proteiner Øvelsesvejledning
 Center for Undervisningsmidler, afdeling København Analyse af proteiner Øvelsesvejledning Formål At separere og analysere proteiner i almindelige fødevarer ved brug af gelelektroforese. Teori Alle dele
Center for Undervisningsmidler, afdeling København Analyse af proteiner Øvelsesvejledning Formål At separere og analysere proteiner i almindelige fødevarer ved brug af gelelektroforese. Teori Alle dele
Biosensor Niveau 1. Teori
 Biosensor Niveau 1 Teori Inden du starter... For at kunne forstå teorien som ligger til grund for en biosensor er det vigtigt at du har styr på nogle generelle mikro/molekyler biologiske principper, begreber
Biosensor Niveau 1 Teori Inden du starter... For at kunne forstå teorien som ligger til grund for en biosensor er det vigtigt at du har styr på nogle generelle mikro/molekyler biologiske principper, begreber
Protokol for genetisk bestemmelse af ABO blodtyper
 Page1 Udarbejdet i samarbejde mellem: Kåre Lehmann, Annabeth Høgh Petersen, Anne Rusborg Nygaard og Jørn M. Clausen Page2 VIGTIGT: SKIFT PIPETTE SPIDSER/TIPS EFTER HVER GANG!! Så undgår du at forurene
Page1 Udarbejdet i samarbejde mellem: Kåre Lehmann, Annabeth Høgh Petersen, Anne Rusborg Nygaard og Jørn M. Clausen Page2 VIGTIGT: SKIFT PIPETTE SPIDSER/TIPS EFTER HVER GANG!! Så undgår du at forurene
Regnskovens hemmeligheder
 Center for Undervisningsmidler, afdeling København Regnskovens hemmeligheder Øvelsesvejledning Formål Et gen for et kræfthelbredende protein er blevet fundet i nogle mystiske blade i regnskoven. Forskere
Center for Undervisningsmidler, afdeling København Regnskovens hemmeligheder Øvelsesvejledning Formål Et gen for et kræfthelbredende protein er blevet fundet i nogle mystiske blade i regnskoven. Forskere
Banan DNA 1/6. Formål: Formålet med øvelsen er at give eleverne mulighed for at se DNA strenge med det blotte øje.
 Banan DNA Formål: Formålet med øvelsen er at give eleverne mulighed for at se DNA strenge med det blotte øje. Baggrundsviden: Om vi er mennesker, dyr eller planter, så har alle organismer DNA i deres celler.
Banan DNA Formål: Formålet med øvelsen er at give eleverne mulighed for at se DNA strenge med det blotte øje. Baggrundsviden: Om vi er mennesker, dyr eller planter, så har alle organismer DNA i deres celler.
Cellekernen (Nucleus) Sebastian Frische Anatomisk Institut
 Cellekernen (Nucleus) Sebastian Frische Anatomisk Institut Cellekernen Cellekernens overordnede struktur kernemembranen/nucleolemma kromatin nucleolus Cellecyklus faser i cellecyklus faser i mitosen Størrelse:
Cellekernen (Nucleus) Sebastian Frische Anatomisk Institut Cellekernen Cellekernens overordnede struktur kernemembranen/nucleolemma kromatin nucleolus Cellecyklus faser i cellecyklus faser i mitosen Størrelse:
Bioteknologi A. Gymnasiale uddannelser. Vejledende opgavesæt 1. Mandag den 31. maj 2010 kl. 9.40-14.40. 5 timers skriftlig prøve
 Vejledende opgavesæt 1 Bioteknologi A Gymnasiale uddannelser 5 timers skriftlig prøve Vejledende opgavesæt 1 Mandag den 31. maj 2010 kl. 9.40-14.40 Side 1 af 8 sider pgave 1. Genmodificeret ris Vitamin
Vejledende opgavesæt 1 Bioteknologi A Gymnasiale uddannelser 5 timers skriftlig prøve Vejledende opgavesæt 1 Mandag den 31. maj 2010 kl. 9.40-14.40 Side 1 af 8 sider pgave 1. Genmodificeret ris Vitamin
Materialeliste: Ready-to-loadTM DNA prøver til elektroforese. Medfølgende reagenser og tilbehør. Egne materialer
 Elevvejledning 1 Materialeliste: Ready-to-loadTM DNA prøver til elektroforese A B C D E F Plasmid DNA (uskåret) Plasmid skåret med Bgl I Plasmid skåret med Eco RI Lambda DNA (uskåret) Lambda DNA skåret
Elevvejledning 1 Materialeliste: Ready-to-loadTM DNA prøver til elektroforese A B C D E F Plasmid DNA (uskåret) Plasmid skåret med Bgl I Plasmid skåret med Eco RI Lambda DNA (uskåret) Lambda DNA skåret
Er der flere farver i sort?
 Er der flere farver i sort? Hvad er kromatografi? Kromatografi benyttes inden for mange forskellige felter og forskningsområder og er en anvendelig og meget benyttet analytisk teknik. Kromatografi bruges
Er der flere farver i sort? Hvad er kromatografi? Kromatografi benyttes inden for mange forskellige felter og forskningsområder og er en anvendelig og meget benyttet analytisk teknik. Kromatografi bruges
Opgave 1 Listeria. mørkviolette bakteriekolonier, se figur 1a. og b. 1. Angiv reaktionstypen for reaktion. 1 vist i figur 1b.
 Opgave 1 Listeria Bakterien Listeria monocytogenes kan være sygdomsfremkaldende for personer, der i forvejen er svækkede. For at identificere Listeria kan man anvende indikative agarplader. Her udnyttes
Opgave 1 Listeria Bakterien Listeria monocytogenes kan være sygdomsfremkaldende for personer, der i forvejen er svækkede. For at identificere Listeria kan man anvende indikative agarplader. Her udnyttes
På grund af reglerne for copyright er det ikke muligt at lægge figurer fra lærebøger på nettet. Derfor har jeg fjernet figurerne fra slides ne, men
 På grund af reglerne for copyright er det ikke muligt at lægge figurer fra lærebøger på nettet. Derfor har jeg fjernet figurerne fra slides ne, men skrevet hvorfra de er taget. De tre bøger, hvorfra illustrationerne
På grund af reglerne for copyright er det ikke muligt at lægge figurer fra lærebøger på nettet. Derfor har jeg fjernet figurerne fra slides ne, men skrevet hvorfra de er taget. De tre bøger, hvorfra illustrationerne
Biotechnology Explorer
 Biotechnology Explorer Oprensning af genomisk DNA fra plantemateriale Manual Katalog nr. 166-5005EDU explorer.bio-rad.com Oversat og bearbejdet af Birgit Sandermann Justesen, Nærum Gymnasium, februar 2009
Biotechnology Explorer Oprensning af genomisk DNA fra plantemateriale Manual Katalog nr. 166-5005EDU explorer.bio-rad.com Oversat og bearbejdet af Birgit Sandermann Justesen, Nærum Gymnasium, februar 2009
Deltagermateriale 43230 OGM. PCR-teknikker 1
 Deltagermateriale 43230 OGM PCR-teknikker 1 POLYMERASE KÆDE REAKTION (PCR) Indholdsfortegnelse 04/02 2 af 36 1. INDLEDNING... 4 DNA's opbygning... 5 DNA-replikation.... 7 2. PCR METODEN... 10 PCR en oversigt...
Deltagermateriale 43230 OGM PCR-teknikker 1 POLYMERASE KÆDE REAKTION (PCR) Indholdsfortegnelse 04/02 2 af 36 1. INDLEDNING... 4 DNA's opbygning... 5 DNA-replikation.... 7 2. PCR METODEN... 10 PCR en oversigt...
Elevvejledning pglo transformation
 Introduktion til transformation Elevvejledning pglo transformation I denne øvelse skal du lære fremgangsmåden ved genetisk transformation. Husk på, at et gen er et stykke DNA, der indeholder informationer
Introduktion til transformation Elevvejledning pglo transformation I denne øvelse skal du lære fremgangsmåden ved genetisk transformation. Husk på, at et gen er et stykke DNA, der indeholder informationer
Struktur og funktion af gener
 Molekylærbiologi og genetik S4, F2008 f Malene Munk Jørgensen Emne: Struktur og funktion af gener Link: undervisningsplanen for S4-molekylærbiologi og genetik MMJ, VI niversity ollege Bioanalytikeruddannelsen
Molekylærbiologi og genetik S4, F2008 f Malene Munk Jørgensen Emne: Struktur og funktion af gener Link: undervisningsplanen for S4-molekylærbiologi og genetik MMJ, VI niversity ollege Bioanalytikeruddannelsen
Danmarks Tekniske Universitet
 Side 1 of 14 Danmarks Tekniske Universitet Skriftlig prøve, den 21/1-2013 Kursus navn: Kursus nr. 27633 Introduktion til Bioinformatik Tilladte hjælpemidler: Alle "Vægtning" Angivet ved de individuelle
Side 1 of 14 Danmarks Tekniske Universitet Skriftlig prøve, den 21/1-2013 Kursus navn: Kursus nr. 27633 Introduktion til Bioinformatik Tilladte hjælpemidler: Alle "Vægtning" Angivet ved de individuelle
at du trænes i at genkende aminosyrer i en simpel proteinstruktur (pentapeptid = lille protein bestående af 5 (penta) aminosyrer)
 Elevvejledning til det Virtuelle Kræftlaboratorium Det Virtuelle Kræftlaboratorium stiller krav til en grundig forståelse af det centrale dogme inden for molekylærbiologien, hvordan DNA oversættes til
Elevvejledning til det Virtuelle Kræftlaboratorium Det Virtuelle Kræftlaboratorium stiller krav til en grundig forståelse af det centrale dogme inden for molekylærbiologien, hvordan DNA oversættes til
Protokol for genetisk bestemmelse af ABO blodtyper
 Page1 Udarbejdet i samarbejde mellem: Kåre Lehmann, Annabeth Høgh Petersen, Anne Rusborg Nygaard og Jørn M. Clausen Page2 VIGTIGT: SKIFT PIPETTE SPIDSER/TIPS EFTER HVER GANG!! Så undgår du at forurene
Page1 Udarbejdet i samarbejde mellem: Kåre Lehmann, Annabeth Høgh Petersen, Anne Rusborg Nygaard og Jørn M. Clausen Page2 VIGTIGT: SKIFT PIPETTE SPIDSER/TIPS EFTER HVER GANG!! Så undgår du at forurene
27611 Eksamen Sommer 2007
 - Side 1 af 10-27611 Eksamen Sommer 2007 Dette sæt indeholder 4 opgaver. En online version af opgavesættet vil være tilgængeligt fra kursets lektionsplan, under selve eksamen (25. Maj 2007 klokken 9:00
- Side 1 af 10-27611 Eksamen Sommer 2007 Dette sæt indeholder 4 opgaver. En online version af opgavesættet vil være tilgængeligt fra kursets lektionsplan, under selve eksamen (25. Maj 2007 klokken 9:00
Kapitel 1 Genteknologi (1/6)
 Kapitel 1 Genteknologi (1/6) Transformation FOTO: SØREN LUNDBERG FORMÅL At isolere plasmider fra plasmidbærende bakterier og overføre dem til andre bakterier. TEORI Transformation er den teknik, hvor generne
Kapitel 1 Genteknologi (1/6) Transformation FOTO: SØREN LUNDBERG FORMÅL At isolere plasmider fra plasmidbærende bakterier og overføre dem til andre bakterier. TEORI Transformation er den teknik, hvor generne
Nye og traditionelle metoder i planteforædling. Søren K. Rasmussen Institut for Plante- og Jordbrugsvidenskab, 27. september 2016
 Nye og traditionelle metoder i planteforædling Søren K. Rasmussen Institut for Plante- og Jordbrugsvidenskab, 27. september 2016 12-10-2016 2 Traditionel forædling Vælge forældrene Krydse forældrene Selektere
Nye og traditionelle metoder i planteforædling Søren K. Rasmussen Institut for Plante- og Jordbrugsvidenskab, 27. september 2016 12-10-2016 2 Traditionel forædling Vælge forældrene Krydse forældrene Selektere
Trin 1: Oprensning af genomisk DNA (DeoxyriboNucleicAcid)
 Trin 1: Oprensning af genomisk DNA (DeoxyriboNucleicAcid) (Denne del tager ca. 3 timer, max 14 prøver) ELEVVEJLEDNING forsøgskasse nr. 1 AAU Oprensning af DNA-prøven foregår gennem fem trin, se figur 1:
Trin 1: Oprensning af genomisk DNA (DeoxyriboNucleicAcid) (Denne del tager ca. 3 timer, max 14 prøver) ELEVVEJLEDNING forsøgskasse nr. 1 AAU Oprensning af DNA-prøven foregår gennem fem trin, se figur 1:
Isolering af DNA fra løg
 Isolering af DNA fra løg Formål: At afprøve en metode til isolering af DNA fra et levende væv. At anvende enzymer.. Indledning: Isolering af DNA fra celler er første trin i mange molekylærbiologiske undersøgelser.
Isolering af DNA fra løg Formål: At afprøve en metode til isolering af DNA fra et levende væv. At anvende enzymer.. Indledning: Isolering af DNA fra celler er første trin i mange molekylærbiologiske undersøgelser.
Biotechnology Explorer. Protein Fingerprinting
 Biotechnology Explorer Protein Fingerprinting Instruktionsmanual Katalognummer 166-0100EDU explorer.bio-rad.com Delene i dette kit er sendt i seperate æsker. Opbevar proteinstandarderne i fryseren, ved
Biotechnology Explorer Protein Fingerprinting Instruktionsmanual Katalognummer 166-0100EDU explorer.bio-rad.com Delene i dette kit er sendt i seperate æsker. Opbevar proteinstandarderne i fryseren, ved
Specifik mutation med nålestiksoperation
 Specifik mutation med nålestiksoperation Mutanter af byg er vigtige i forskningen og benyttes også, når der forædles nye sorter til dyrkning. Hidtil har det kun været muligt at inducere mutationer tilfældige
Specifik mutation med nålestiksoperation Mutanter af byg er vigtige i forskningen og benyttes også, når der forædles nye sorter til dyrkning. Hidtil har det kun været muligt at inducere mutationer tilfældige
BIOLOGI HØJT NIVEAU. Mandag den 13. august 2001 kl
 STUDENTEREKSAMEN AUGUST 2001 2001-6-2 BIOLOGI HØJT NIVEAU Mandag den 13. august 2001 kl. 9.00-14.00 Af de store opgaver 1 og 2 må kun den ene besvares. Af de små opgaver 3, 4, 5 og 6 må kun to besvares.
STUDENTEREKSAMEN AUGUST 2001 2001-6-2 BIOLOGI HØJT NIVEAU Mandag den 13. august 2001 kl. 9.00-14.00 Af de store opgaver 1 og 2 må kun den ene besvares. Af de små opgaver 3, 4, 5 og 6 må kun to besvares.
Klip-og-kopier DNA: reparér mutationer med 'genom-redigering' DNA, RNA og protein
 Forskningsnyheder om Huntingtons Sygdom På hverdagssprog Skrevet af forskere. Til det globale HS-fællesskab Klip-og-kopier DNA: reparér mutationer med 'genom-redigering' Forskere kan lave præcise ændringer
Forskningsnyheder om Huntingtons Sygdom På hverdagssprog Skrevet af forskere. Til det globale HS-fællesskab Klip-og-kopier DNA: reparér mutationer med 'genom-redigering' Forskere kan lave præcise ændringer
Bioteknologi A. Studentereksamen. Af opgaverne 1 og 2 skal begge opgaver besvares. Af opgaverne 3 og 4 skal en og kun en af opgaverne besvares.
 Bioteknologi A Studentereksamen Af opgaverne 1 og 2 skal begge opgaver besvares. Af opgaverne 3 og 4 skal en og kun en af opgaverne besvares. frs111-btk/a-31052011 Tirsdag den 31. maj 2011 kl. 9.00-14.00
Bioteknologi A Studentereksamen Af opgaverne 1 og 2 skal begge opgaver besvares. Af opgaverne 3 og 4 skal en og kun en af opgaverne besvares. frs111-btk/a-31052011 Tirsdag den 31. maj 2011 kl. 9.00-14.00
Danmarks Tekniske Universitet
 Side 1 of 13 Danmarks Tekniske Universitet Skriftlig prøve, den 22/2-2013 Kursus navn: Introduktion til SystemBiologi Tilladte hjælpemidler: Alle "Vægtning" Angivet ved de individuelle opgaver. Kursusansvarlig
Side 1 of 13 Danmarks Tekniske Universitet Skriftlig prøve, den 22/2-2013 Kursus navn: Introduktion til SystemBiologi Tilladte hjælpemidler: Alle "Vægtning" Angivet ved de individuelle opgaver. Kursusansvarlig
1. Hvad er kræft, og hvorfor opstår sygdommen?
 1. Hvad er kræft, og hvorfor opstår sygdommen? Dette kapitel fortæller om, cellen, kroppens byggesten hvad der sker i cellen, når kræft opstår? årsager til kræft Alle levende organismer består af celler.
1. Hvad er kræft, og hvorfor opstår sygdommen? Dette kapitel fortæller om, cellen, kroppens byggesten hvad der sker i cellen, når kræft opstår? årsager til kræft Alle levende organismer består af celler.
August 2016 Outbreak Copyright: Lise Junker Nielsen & Allan Haurballe. Foto og illustrationer: Colourbox Kontakt:
 OutBreak 1 August 2016 Outbreak Copyright: Lise Junker Nielsen & Allan Haurballe. Foto og illustrationer: Colourbox Kontakt: lisej@bmb.sdu.dk Undervisningsforløbet og tilhørende forsøg udføres på eget
OutBreak 1 August 2016 Outbreak Copyright: Lise Junker Nielsen & Allan Haurballe. Foto og illustrationer: Colourbox Kontakt: lisej@bmb.sdu.dk Undervisningsforløbet og tilhørende forsøg udføres på eget
Teknikken er egentlig meget simpel og ganske godt illustreret på animationen shell 4-5.
 Fysikken bag Massespektrometri (Time Of Flight) Denne note belyser kort fysikken bag Time Of Flight-massespektrometeret, og desorptionsmetoden til frembringelsen af ioner fra vævsprøver som er indlejret
Fysikken bag Massespektrometri (Time Of Flight) Denne note belyser kort fysikken bag Time Of Flight-massespektrometeret, og desorptionsmetoden til frembringelsen af ioner fra vævsprøver som er indlejret
Bioteknologi A Kommentarer til opgaveformuleringer i opgavesættet 31. maj 2011
 Bioteknologi A Kommentarer til opgaveformuleringer i opgavesættet 31. maj 2011 Spørgsmålene i opgavesættene stilles som udgangspunkt i den betydning der fremgår af listen over typeord, som findes på ministeriets
Bioteknologi A Kommentarer til opgaveformuleringer i opgavesættet 31. maj 2011 Spørgsmålene i opgavesættene stilles som udgangspunkt i den betydning der fremgår af listen over typeord, som findes på ministeriets
Drikkevandssensorer 2016
 Drikkevandssensorer 2016 Teknisk gennemgang af målemetoder Vandhuset, 27. januar 2016 Loren Ramsay, VIA Engineering Monitering Vi vil gerne monitere for bakterier i vores drikkevand. Men kan vi blive mere
Drikkevandssensorer 2016 Teknisk gennemgang af målemetoder Vandhuset, 27. januar 2016 Loren Ramsay, VIA Engineering Monitering Vi vil gerne monitere for bakterier i vores drikkevand. Men kan vi blive mere
Hvad er så vigtigt ved målinger?
 Forskningsnyheder om Huntingtons Sygdom På hverdagssprog Skrevet af forskere. Til det globale HS-fællesskab Spændende opdagelse i blodceller fra patienter med Huntingtons Sygdom Mængden af huntingtinprotein
Forskningsnyheder om Huntingtons Sygdom På hverdagssprog Skrevet af forskere. Til det globale HS-fællesskab Spændende opdagelse i blodceller fra patienter med Huntingtons Sygdom Mængden af huntingtinprotein
BIOLOGI A-NIVEAU NY ORDNING. Tirsdag den 19. august 2008. Kl. 09.00 14.00 STX082-BIA STUDENTEREKSAMEN AUGUST 2008
 STUDENTEREKSAMEN AUGUST 2008 BIOLOGI A-NIVEAU Tirsdag den 19. august 2008 NY ORDNING Kl. 09.00 14.00 Af opgaverne 1, 2, 3 og 4 skal tre og kun tre af opgaverne besvares STX082-BIA Undervisningsministeriet
STUDENTEREKSAMEN AUGUST 2008 BIOLOGI A-NIVEAU Tirsdag den 19. august 2008 NY ORDNING Kl. 09.00 14.00 Af opgaverne 1, 2, 3 og 4 skal tre og kun tre af opgaverne besvares STX082-BIA Undervisningsministeriet
Genekspression. GENEKSPRESSION side 1
 Genekspression 2006 GENEKSPRESSION side 1 Indholdsfortegnelse Kapitel 1... 3 Proteinsyntese... 3 Regulering af gener i E. coli.... 8 Transskription hos eukaryoter... 14 Eukaryot genregulering... 16 Efterbehandlig
Genekspression 2006 GENEKSPRESSION side 1 Indholdsfortegnelse Kapitel 1... 3 Proteinsyntese... 3 Regulering af gener i E. coli.... 8 Transskription hos eukaryoter... 14 Eukaryot genregulering... 16 Efterbehandlig
Protein syntese. return
 Protein syntese. I artiklen redegøres for principperne i, hvordan octapeptidet SCHTFGDI kan syntetiseres. Som yderligere illustration heraf kan peptidet opbygges og visualiseres i Chem3D-Pro. Herved kan
Protein syntese. I artiklen redegøres for principperne i, hvordan octapeptidet SCHTFGDI kan syntetiseres. Som yderligere illustration heraf kan peptidet opbygges og visualiseres i Chem3D-Pro. Herved kan
Livets molekylære kode
 4. ÅR R. 2 / 2006 Livets molekylære kode -kemi og biologi mødes D livets molekylære kode D findes i alle levende organismer og indeholder koden til organismen informationer, der afgør om organismen er
4. ÅR R. 2 / 2006 Livets molekylære kode -kemi og biologi mødes D livets molekylære kode D findes i alle levende organismer og indeholder koden til organismen informationer, der afgør om organismen er
Organismer inddeles i tre fundamentale stofomsætningstyper:
 Stofskiftetyper Organismer inddeles i tre fundamentale stofomsætningstyper: autotrofe organismer: organismer som opbygger organisk stof ved fotosyntese (eller i nogle tilfælde kemosyntese); de kræver foruden
Stofskiftetyper Organismer inddeles i tre fundamentale stofomsætningstyper: autotrofe organismer: organismer som opbygger organisk stof ved fotosyntese (eller i nogle tilfælde kemosyntese); de kræver foruden
TEST 1. Almen Cellebiologi 2004/2005
 TEST 1 Almen Cellebiologi 2004/2005 Hvilken af følgende udsagn er forkerte? a) kun planteceller har en cellevæg b) planteceller har microtubuli c) planteceller har chloroplaster d) planteceller har actin
TEST 1 Almen Cellebiologi 2004/2005 Hvilken af følgende udsagn er forkerte? a) kun planteceller har en cellevæg b) planteceller har microtubuli c) planteceller har chloroplaster d) planteceller har actin
Dette er en kladde til et genoptryk af Eksperimentel Genteknologi fra 1991. Ideer, rettelser og forslag modtages gerne. Kh Claudia.
 Transformation af E.coli K 12 Version 3. marts 2009 (C) Claudia Girnth-Diamba og Bjørn Fahnøe Dette er en kladde til et genoptryk af Eksperimentel Genteknologi fra 1991. Ideer, rettelser og forslag modtages
Transformation af E.coli K 12 Version 3. marts 2009 (C) Claudia Girnth-Diamba og Bjørn Fahnøe Dette er en kladde til et genoptryk af Eksperimentel Genteknologi fra 1991. Ideer, rettelser og forslag modtages
Side%1%af%14% Eksamen: Bioinformatik It og Sundhed 27 Jan 2011 kl 9-13
 Side1af14 Eksamen: Bioinformatik It og Sundhed 27 Jan 2011 kl 9-13 Navn: Studie nummer: Dette eksamenssæt vil også kunne ses som en pdf fil nederst på kursus-hjemmesiden udfor den sidste dag d. 27 Jan
Side1af14 Eksamen: Bioinformatik It og Sundhed 27 Jan 2011 kl 9-13 Navn: Studie nummer: Dette eksamenssæt vil også kunne ses som en pdf fil nederst på kursus-hjemmesiden udfor den sidste dag d. 27 Jan
1 Idékatalog til øvelser: Bioanalytikeruddannelsen UCSJ, Parkvej 190, 4700 Næstved
 1 Idékatalog til øvelser: Bioanalytikeruddannelsen UCSJ, Parkvej 190, 2 Idékatalog til øvelser: Bioanalytikeruddannelsen UCSJ, Parkvej 190, Indhold Realtime PCR på β-actin... 3 CSI og retsgenetik... 3
1 Idékatalog til øvelser: Bioanalytikeruddannelsen UCSJ, Parkvej 190, 2 Idékatalog til øvelser: Bioanalytikeruddannelsen UCSJ, Parkvej 190, Indhold Realtime PCR på β-actin... 3 CSI og retsgenetik... 3
Eksamensspørgsmål til BiB biologi B 2015
 Eksamensspørgsmål til BiB biologi B 2015 Med udgangspunkt i de udleverede bilag og temaet evolution skal du: 1. Redegøre for nogle forskellige teorier om evolution, herunder begrebet selektion. 2. Analysere
Eksamensspørgsmål til BiB biologi B 2015 Med udgangspunkt i de udleverede bilag og temaet evolution skal du: 1. Redegøre for nogle forskellige teorier om evolution, herunder begrebet selektion. 2. Analysere
Proteinkarakterisering ved massespektrometri
 Proteinkarakterisering ved massespektrometri - fra fødevarer til kræftforskning Af Mette Rindom Nørrelykke, Hans Peter Sørensen og Hans Christian Beck, Teknologisk Institut Viden om proteiner er vigtig
Proteinkarakterisering ved massespektrometri - fra fødevarer til kræftforskning Af Mette Rindom Nørrelykke, Hans Peter Sørensen og Hans Christian Beck, Teknologisk Institut Viden om proteiner er vigtig
Bestemmelse af koffein i cola
 Bestemmelse af koffein i cola 1,3,7-trimethylxanthine Koffein i læskedrikke Læs følgende links, hvor der blandt andet står nogle informationer om koffein og regler for hvor meget koffein, der må være i
Bestemmelse af koffein i cola 1,3,7-trimethylxanthine Koffein i læskedrikke Læs følgende links, hvor der blandt andet står nogle informationer om koffein og regler for hvor meget koffein, der må være i
Proteiner: en introduktion. Modul 1; F13 Rolf Andersen, 18/2-2013
 Proteiner: en introduktion Modul 1; F13 Rolf Andersen, 18/2-2013 4 facts om proteiner Proteiner udgør én af de vigtigste stofgrupper i vores organisme; de varetager en lang række forskellige funktioner.
Proteiner: en introduktion Modul 1; F13 Rolf Andersen, 18/2-2013 4 facts om proteiner Proteiner udgør én af de vigtigste stofgrupper i vores organisme; de varetager en lang række forskellige funktioner.
Eksamen i. Cellebiologi (kandidatdelen): Cellebiologi - Cellers struktur og funktion - Membranbiokemi - Cellulær signaltransduktion
 Eksamen i Cellebiologi (kandidatdelen): Cellebiologi - Cellers struktur og funktion - Membranbiokemi - Cellulær signaltransduktion Opgavesættet består af 5 sider inklusive denne forside. Sættet består
Eksamen i Cellebiologi (kandidatdelen): Cellebiologi - Cellers struktur og funktion - Membranbiokemi - Cellulær signaltransduktion Opgavesættet består af 5 sider inklusive denne forside. Sættet består
BIOTEKNOLOGI HØJT NIVEAU
 STUDETEREKSAME 2006 2006-BT-2 BIOTEKOLOGI HØJT IVEAU Onsdag den 16. august 2006 kl. 9.00 14.00 Sættet består af 1 stor og 2 små opgaver. Alle hjælpemidler tilladt. STOR OPGAVE 1. Myoglobin A. Den røde
STUDETEREKSAME 2006 2006-BT-2 BIOTEKOLOGI HØJT IVEAU Onsdag den 16. august 2006 kl. 9.00 14.00 Sættet består af 1 stor og 2 små opgaver. Alle hjælpemidler tilladt. STOR OPGAVE 1. Myoglobin A. Den røde
NDRG2 s Interaction with Api5
 Naturvidenskabeligt Basisstudium Roskilde Universitet 2. semesterprojekt Juni 2012 Hus 13.1 NDRG2 s Interaction with Api5 NDRG2 s interaktion med Api5 Gruppe 12: Freya Armstrong Annaleigh Ohrt Fehler Cecilie
Naturvidenskabeligt Basisstudium Roskilde Universitet 2. semesterprojekt Juni 2012 Hus 13.1 NDRG2 s Interaction with Api5 NDRG2 s interaktion med Api5 Gruppe 12: Freya Armstrong Annaleigh Ohrt Fehler Cecilie
BIOLOGI A-NIVEAU NY ORDNING. Tirsdag den 20. maj 2008. Kl. 09.00 14.00 STX081-BIA STUDENTEREKSAMEN MAJ 2008
 STUDENTEREKSAMEN MAJ 2008 BIOLOGI A-NIVEAU Tirsdag den 20. maj 2008 NY ORDNING Kl. 09.00 14.00 Af opgaverne 1, 2, 3 og 4 skal tre og kun tre af opgaverne besvares STX081-BIA Undervisningsministeriet Side
STUDENTEREKSAMEN MAJ 2008 BIOLOGI A-NIVEAU Tirsdag den 20. maj 2008 NY ORDNING Kl. 09.00 14.00 Af opgaverne 1, 2, 3 og 4 skal tre og kun tre af opgaverne besvares STX081-BIA Undervisningsministeriet Side
Side 1 of 11. Kursus navn: Kursus nr Introduktion til Bioinformatik
 Side 1 of 11 Danmarks Tekniske Universitet Skriftlig prøve, den 22/1-2015 Kursus navn: Kursus nr. 27633 Introduktion til Bioinformatik Tilladte hjælpemidler: Alle "Vægtning" Angivet ved de individuelle
Side 1 of 11 Danmarks Tekniske Universitet Skriftlig prøve, den 22/1-2015 Kursus navn: Kursus nr. 27633 Introduktion til Bioinformatik Tilladte hjælpemidler: Alle "Vægtning" Angivet ved de individuelle
Kemi Kulhydrater og protein
 Kemi Kulhydrater og protein Formål: Formålet med forsøget er at vise hvordan man kan påvise protein, fedtstof, simple sukkerarter eller stivelse i forskellige fødevarer. Samtidig kan man få en fornemmelse
Kemi Kulhydrater og protein Formål: Formålet med forsøget er at vise hvordan man kan påvise protein, fedtstof, simple sukkerarter eller stivelse i forskellige fødevarer. Samtidig kan man få en fornemmelse
# Problemet med genetisk ustabilitet
 Forskningsnyheder om Huntingtons Sygdom På hverdagssprog Skrevet af forskere. Til det globale HS-fællesskab Et DNA-reparerende protein ændrer stabiliteten af lange CAG-områder i det muterede gen for Huntingtons
Forskningsnyheder om Huntingtons Sygdom På hverdagssprog Skrevet af forskere. Til det globale HS-fællesskab Et DNA-reparerende protein ændrer stabiliteten af lange CAG-områder i det muterede gen for Huntingtons
Mitokondrier og oxidativt stress
 Forskningsnyheder om Huntingtons Sygdom På hverdagssprog Skrevet af forskere. Til det globale HS-fællesskab At gå målrettet mod oxidativ stress i Huntingtons Sygdom Skade på celler skabt af oxidativt stress
Forskningsnyheder om Huntingtons Sygdom På hverdagssprog Skrevet af forskere. Til det globale HS-fællesskab At gå målrettet mod oxidativ stress i Huntingtons Sygdom Skade på celler skabt af oxidativt stress
Øvelser 10. KlasseCenter Vesthimmerland Kaj Mikkelsen
 Indholdsfortegnelse Indholdsfortegnelse... 1 Bygning af et glucosemolekyle... 2 Bygning af et poly- sakkarid.... 3 Påvisning af glukose (1)... 4 Påvisning af glucose (2)... 5 Påvisning af disakkarider....
Indholdsfortegnelse Indholdsfortegnelse... 1 Bygning af et glucosemolekyle... 2 Bygning af et poly- sakkarid.... 3 Påvisning af glukose (1)... 4 Påvisning af glucose (2)... 5 Påvisning af disakkarider....
Spm. 1.: Hvis den totale koncentration af monomer betegnes med CT hvad er så sammenhængen mellem CT, [D] og [M]?
![Spm. 1.: Hvis den totale koncentration af monomer betegnes med CT hvad er så sammenhængen mellem CT, [D] og [M]? Spm. 1.: Hvis den totale koncentration af monomer betegnes med CT hvad er så sammenhængen mellem CT, [D] og [M]?](/thumbs/32/15386991.jpg) DNA-smeltetemperaturbestemmelse KemiF2-2008 DNA-smeltetemperaturbestemmelse Introduktion Oligonucleotider er ofte benyttet til at holde nanopartikler sammen med hinanden. Den ene enkeltstreng er kovalent
DNA-smeltetemperaturbestemmelse KemiF2-2008 DNA-smeltetemperaturbestemmelse Introduktion Oligonucleotider er ofte benyttet til at holde nanopartikler sammen med hinanden. Den ene enkeltstreng er kovalent
KRIMINALDETEKTIVEN GERNINGSSTEDETS GÅDE PCR
 KRIMINALDETEKTIVEN GERNINGSSTEDETS GÅDE Elevvejledning PCR PCR trin for trin PCR involverer en række gentagne cykler, der hver består af følgende tre trin: Denaturering, primer påsætning og forlængelse
KRIMINALDETEKTIVEN GERNINGSSTEDETS GÅDE Elevvejledning PCR PCR trin for trin PCR involverer en række gentagne cykler, der hver består af følgende tre trin: Denaturering, primer påsætning og forlængelse
Eukaryote mrna Processering
 Eukaryote mrna Processering Splicing Capping Poly-adenylering Post-transkriptionel regulering af genexpression Simpel prokaryotisk gen-ekspression Alt RNA bliver syntetiseret af en enkelt RNA-polymerase
Eukaryote mrna Processering Splicing Capping Poly-adenylering Post-transkriptionel regulering af genexpression Simpel prokaryotisk gen-ekspression Alt RNA bliver syntetiseret af en enkelt RNA-polymerase
Re- eksamen Det hæmatologiske system og immunsystemet. Bacheloruddannelsen i Medicin/Medicin med industriel specialisering
 Studienummer: 1 Re- eksamen 2012 Titel på kursus: Uddannelse: Semester: Det hæmatologiske system og immunsystemet Bacheloruddannelsen i Medicin/Medicin med industriel specialisering 2. semester Eksamensdato:
Studienummer: 1 Re- eksamen 2012 Titel på kursus: Uddannelse: Semester: Det hæmatologiske system og immunsystemet Bacheloruddannelsen i Medicin/Medicin med industriel specialisering 2. semester Eksamensdato:
Laboratorieprotokol for manuel isolering af DNA fra 0,5 ml prøve
 Laboratorieprotokol for manuel isolering af DNA fra 0,5 ml prøve Til isolering af genomisk DNA fra indsamlingssætserierne Oragene - og ORAcollect. Du kan finde flere sprog og protokoller på vores webside
Laboratorieprotokol for manuel isolering af DNA fra 0,5 ml prøve Til isolering af genomisk DNA fra indsamlingssætserierne Oragene - og ORAcollect. Du kan finde flere sprog og protokoller på vores webside
Test Canvas: Eksamen i BMB502 Januar 2012
 BMB502, Enzymer og membraner, efterår 11. f Tests, Surveys and Pools Tests Test Canvas : Eksamen i BMB502 Januar 2012 Edit Mode is: Test Canvas: Eksamen i BMB502 Januar 2012 Create Reuse Upload s Settings
BMB502, Enzymer og membraner, efterår 11. f Tests, Surveys and Pools Tests Test Canvas : Eksamen i BMB502 Januar 2012 Edit Mode is: Test Canvas: Eksamen i BMB502 Januar 2012 Create Reuse Upload s Settings
Protein identifikation ved hjælp af Matrix- Assisted Laser Desorption Masse Spektrometri (MALDI-MS).
 Teori til elevforløb på Institut for Biokemi og Molekylærbiologi på Syddansk Universitet: Protein identifikation ved hjælp af Matrix- Assisted Laser Desorption Masse Spektrometri (MALDI-MS). Peter Windfeldt
Teori til elevforløb på Institut for Biokemi og Molekylærbiologi på Syddansk Universitet: Protein identifikation ved hjælp af Matrix- Assisted Laser Desorption Masse Spektrometri (MALDI-MS). Peter Windfeldt
